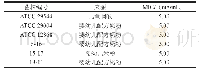
《表1 Cronobacter PubMLST数据库中的克罗诺杆菌菌株统计》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《多位点序列分析方法在克罗诺杆菌属菌株溯源分析上的应用》
注:a为序列类型(Sequence type)。b为占物种总数的百分比(Percentage of speciestotal)。c为占总属的百分比(Percentage of genus total)。
目前Cronobacter PubMLST基因组和序列定义数据库(http://pubmlst.org/cronobacter/)已包含超过2400多株菌株分离信息,其中C.sakazakii(1 774株)和C.malonaticus(295株)为该属的优势菌种(表1)。通过将7-loci MLST应用于2 438株菌进行分析,共揭示了591种可定义的序列型(sequence type,ST),其中克诺罗杆菌C.sakazakii ST1(280株)、ST4(334株)、ST13(67株)和C.malonaticus ST7(78株)为主要序列型(表2)。截止2019年3月20日,数据库中已有623株菌完成全基因组测序,包括C.sakazakii(438株)、C.malonaticus(82株)、C.dublinensis(56株)、C.turicensis(23株)、C.muytjensii(13株)C.universalis(9株)和C.condimenti(2株)。数据库中最早的分离株为1950年分离自英国奶粉中的C.sakazakii,所有菌株共分离自全球40多个国家和地区。多数菌株分离自食品及配料当中,C.sakazakii和C.malonaticus与临床及婴幼儿配方奶粉密切相关。C.sakazakii和C.malonaticus中主要序列类型的国家来源统计结果显示,多数菌株分离自中国和美国(表1、2)。上述结果表明,由于MLST所针对的七个基因位点在基因组总量中占比较小,导致同一ST型中包含地理来源,分离时间,宿主等不相关的菌株,而对菌株溯源结果适得其反。
| 图表编号 | XD00110689200 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.09.25 |
| 作者 | 闫瑞、杨捷琳、陈翠玲、钮冰、徐之雯、蒋原 |
| 绘制单位 | 上海大学生命科学学院、上海出入境检验检疫局、上海出入境检验检疫局、上海大学生命科学学院、上海出入境检验检疫局、上海大学生命科学学院、上海出入境检验检疫局、上海出入境检验检疫局 |
| 更多格式 | 高清、无水印(增值服务) |