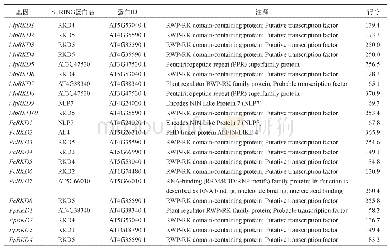
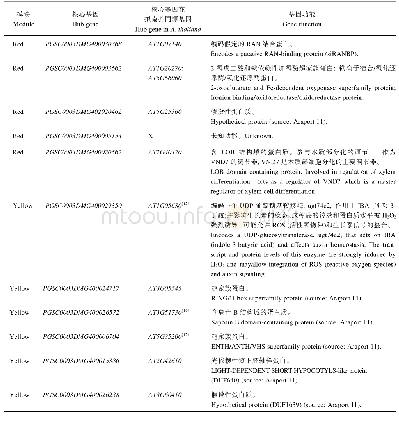
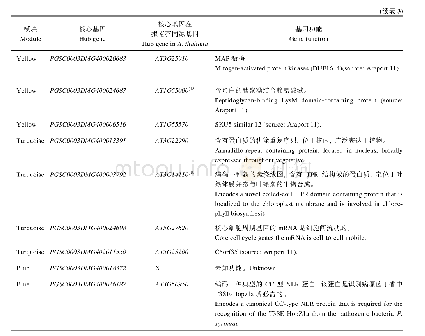
《表2 模块核心基因功能注释》
水稻干旱胁迫、冷胁迫、盐胁迫处理的100个转录组数据来源于NCBI的SRA(Sequence Read Archive)数据库(附表2)。首先利用SRAtoolkit[9]软件的fastdump将SRA格式文件转换为序列文件fastq,利用FastQC[10]软件对原始测序数据进行质量评估,紧接着用Trimmonatic[11]软件去除接头、过滤低质量的reads,得到clean data。利用Hisat2[12]软件将clean data比对到水稻基因组。水稻基因组序列和注释信息下载自Rice Genome Annotation Project(http://rice.plantbiology.msu.edu)数据库。用featureCounts[13]获得数据的reads计数,之后根据基因表达的TPM值(transcripts permillion)公式,编写计算TPM值的R语言代码,构建基因表达矩阵,R语言代码如下:
| 图表编号 | XD0078535900 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.09.12 |
| 作者 | 李旭凯、李任建、张宝俊 |
| 绘制单位 | 山西农业大学生命科学学院、山西农业大学农学院、山西农业大学农学院 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式