《表2 GenBank上获取的FSHR序列信息》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《林麝(Moschus moschiferus)FSHR基因的克隆及进化分析》
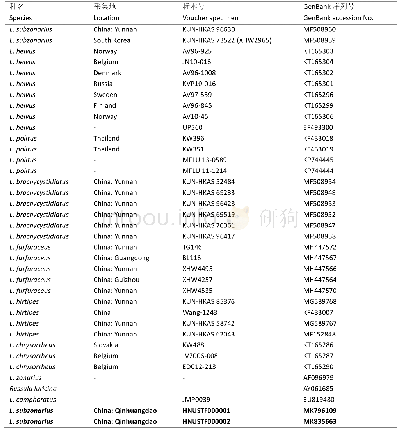
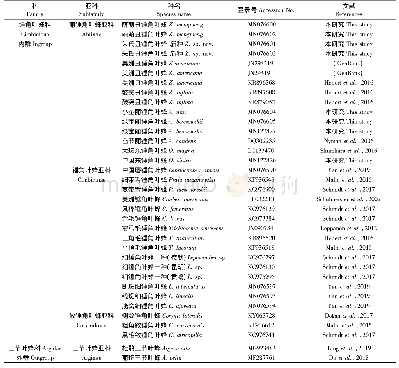
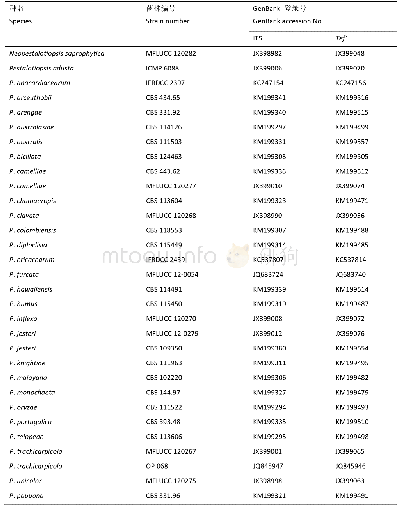
序列校对使用Chmmosoma 1.62、DNAStar 7.2等软件。同源相似性及蛋白质的预测采用DNAMAN软件[15]。疏水性分析使用ExpASy protenomics服务器(https://www.expasy.org/proteomics)中的protscale进行预测分析。采用在线分析软件HNN secondary structure predictionmethod(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_hnn.html)对林麝FSHR基因蛋白的二级结构进行预测。采用MEGA5.0软件分析序列特征,计算序列间碱基对的差异信息[15]。从GenBank中下载了12种动物的FSHR基因序列(表2),用MEGA5.0构建分子进化树,采用的方法为NJ(neighbor-joining)及ME(minimum evolution)法,分支置信度采用自展(Bootstrap)检验法进行评价,自展检验重复次数为1 000次。
| 图表编号 | XD0056481200 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.04.28 |
| 作者 | 欧阳菠、张明、蔡永华、杨营、郑程莉、程建国、周明、李彪、钟赟、徐丰奕、杨建东 |
| 绘制单位 | 四川农业大学动物科技学院、四川农业大学动物科技学院、四川养麝研究所、四川养麝研究所、四川养麝研究所、四川养麝研究所、四川农业大学动物科技学院、四川农业大学动物科技学院、四川农业大学动物科技学院、四川农业大学动物科技学院、四川农业大学动物科技学院 |
| 更多格式 | 高清、无水印(增值服务) |