《表1 GenBank已发表序列信息》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《猪线粒体控制区微卫星(mtMs)变异及其分布特征》
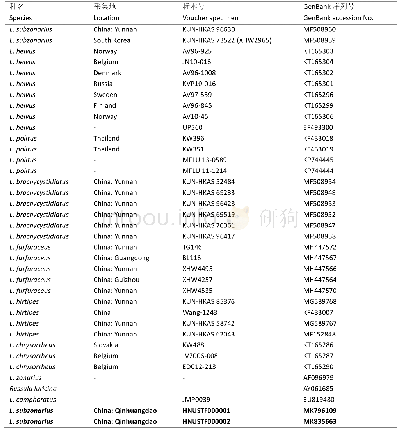
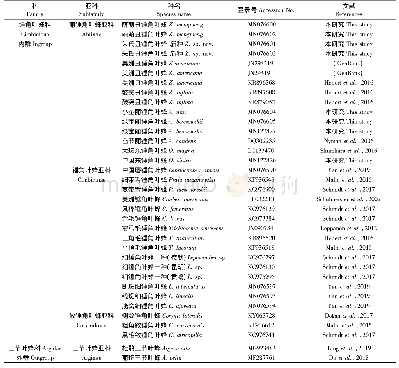
通过对GenBank(https://www.ncbi.nlm.nih.gov/genbank/)中地方猪种、选育猪种和野猪210个个体的mtDNA D-loop区比对分析,表明核苷酸序列长度存在较大程度差异,介于1 064 bp(AY879772,韩国地方猪)和1 505 bp(EU117375,西班牙野猪)之间,造成这种差异的原因在于D-loop中重复单元“5'-TGC GTACACG-3'”重复次数不同(表1)。为进一步分析结构特征,对中国4个藏猪群体、八眉猪、烟台黑猪及引入猪种杜洛克、大白和长白猪164个个体mtDNA D-loop核苷酸序列进行了分析,结果表明,“5'-TGCG TACACG-3'”并不是唯一的微卫星片段,存在多个变异形式,进而形成了复合mtMs结构。分析发现,“5'-TGCGTACACG-3'”(RA)是mtMs重复单元的核心序列,其中第2位、第4位和第10位上的“G”突变为“A”和第3位“C”突变为“T”而产生了相应的变异序列(RA-RG)(表2) 。长度分析表明:mtDNA D-loop序列碱基长度差异数在10的倍数水平。序列比对结果显示,mtMs区域最短片段为30 bp,最长470 bp,均因重复单元重复次数差异而产生,即个体间mtMs的重复单元数介于3~47之间。有64.44%(241)的个体mtMs区域是由核心序列RA的组成的单一重复(重复次数3~47),而35.56%(133)的个体是由核心序列及其变异结构相互交错组成的复合mtMs结构(重复次数7~30)。
| 图表编号 | XD0062463000 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.07.25 |
| 作者 | 蔡原、姜天团、杨巧丽、王川、赵生国 |
| 绘制单位 | 甘肃农业大学动物科学技术学院、甘肃农业大学动物科学技术学院、甘肃农业大学动物科学技术学院、甘肃农业大学动物医学院、甘肃农业大学动物科学技术学院 |
| 更多格式 | 高清、无水印(增值服务) |