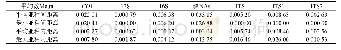
《表7 Bl CCo AOMT基因在群体内的核苷酸变异》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《光皮桦BlCCoAOMT基因的克隆、表达及单核苷酸变异分析》
注:πtot:整个基因区域单核苷酸多样性;πsil:非翻译区和内含子区域的单核苷酸多样性;πs:编码区同义突变单核苷酸多样性;πn:编码区内非同义突变多样性。Tajima's D和Fu and Li's D为两种不同检验中性进化的方法得到的D值。Note:πtot:Average nucleotide diversity in full ge
核苷酸多样性是衡量一个群体之中遗传多样性的重要参数。多样性数值越小,代表着群体的多样性程度越低[26]。由表7可知,利用119个SNP位点计算得到多样性指数πtot、πsil、πs、πn,显示这些参数在4个群体间都存在一定差异,但均未达到显著水平,表明在不同群体间BlCCoAOMT基因的分化程度无显著差异。所有4个群体的非同义突变多样性指数πn均小于同义突变πs,说明在光皮桦的进化过程中,BlCCoAOMT基因主要受到纯化的选择压力。利用DnaSP 5.0软件对4个群体进行了Tajima's D和Fu and Li's D的中性检验,发现福建尤溪、广西龙胜和江西龙南3个群体Tajima's D均为负值,表明这些群体中存在较多低频率的等位基因,仅浙江临安群体Tajima's D为正值,暗示中等频率的等位基因占主导。福建尤溪和广西龙胜2个群体Fu and Li's D均为负值,表明进化过程中存在较多变异,多样性丰富;而江西龙南和浙江临安为正值,表明进化过程中,正向选择占主导地位[27-28]。
| 图表编号 | XD0052363300 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.05.10 |
| 作者 | 俞子承、倪飞、江成、黄华宏、林二培、童再康 |
| 绘制单位 | 浙江农林大学亚热带森林培育国家重点实验室、浙江农林大学亚热带森林培育国家重点实验室、浙江农林大学亚热带森林培育国家重点实验室、浙江农林大学亚热带森林培育国家重点实验室、浙江农林大学亚热带森林培育国家重点实验室、浙江农林大学亚热带森林培育国家重点实验室 |
| 更多格式 | 高清、无水印(增值服务) |

![表1.鼠衣原体在C57BL/6J小鼠肠道内的生长情况[log10(xs)]](http://bookimg.mtoou.info/tubiao/gif/WSXB202004011_03600.gif)