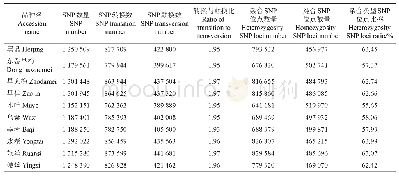
《表3 样品检测的SNP统计》
SNP的检测主要使用GATK软件工具包实现,以保证检测得到的SNP的准确性并得到最终的SNP位点集。主要检测过程首先使用GATK进行In Del Realignment,即对存在插入缺失比对结果附近的位点进行局部重新比对,校正由于插入缺失引起的比对结果错误;其次使用GATK和Samtools进行变异检测,主要包括SNP和In Del;第三根据GATK和samtools分别得到的变异中将位点一致的部分作为最终的变异位点用于后续分析。具体流程参考GATK官方网站的Best Practice。所有样品的SNP统计信息见表3。
| 图表编号 | XD0028197500 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.03.01 |
| 作者 | 庞贞武、刘鑫、孙雪阳、兰俊、陈德文 |
| 绘制单位 | 广西国有东门林场、广西国有东门林场、广西国有东门林场、广西国有东门林场、广西国有东门林场 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式