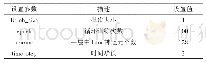
《表1 CDOCKER、LIBDOCK和GOLD对接中的参数设置 (未列出的参数均使用程序的默认值)》
我们采用分子对接的虚拟筛选方式,利用D i s c o v e r y S t u d i o(D S)2.5和G O L D 3.0两个软件进行受体与配体间的分子对接筛选(DS中仅应用“CDOCKER”与“LIBCOCK”两个对接模块)。将预处理后的天然产物化合物分子构象,基于CDOCKER的柔性对接方法、LIBCOCK的刚性对接方法和GOLD 3.0的遗传算法进行受体与配体的分子对接虚拟筛选,将分子对接得到复合物结构作为初步虚拟筛选的结果,而后进行复合物的能量优化与进一步的筛选。各对接方法的简要流程如下,具体参数设置见表1。
| 图表编号 | XD0022927000 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2018.03.01 |
| 作者 | 冯迪、刘湲、孙影、郑珩 |
| 绘制单位 | 中国药科大学生命科学与技术学院、中国药科大学生命科学与技术学院、中国药科大学生命科学与技术学院、中国药科大学生命科学与技术学院 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式