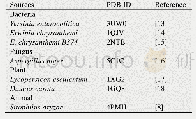
《表3.PDB数据库中已发布的CE酶晶体结构》
尽管纤维二糖差向异构酶催化乳糖的效率较高,但仍然存在诸多问题;首先是副产物依匹乳糖的比例过高,尽管依匹乳糖本身也是一种人体益生元[53–56],但各国药典对其含量均有严格限制。蛋白质工程改造是解决这一难题的途径,其中定向进化显示了这方面的优势。Shen等通过定向进化筛选到了五位点突变体R5M/I52V/A12S/K328I/F231L,该突变体并不产生副产物依匹乳糖[40],说明定向进化是非常有效的纤维二糖差向异构酶改造方法。此外,根据PDB数据库中已发布的CE酶晶体结构数据,通过分子同源建模和理性设计的方法,也可以对CE酶进行有效改造。截止目前,PDB数据库共有5种CE酶的晶体结构(表3),已报道的CE酶多为单体酶,分子量43–47 kDa。分析多种来源的CE酶表明氨基酸序列相似度在35%–50%,氨基酸序列保守区包括No.50–60、No.180–200、No.240–260、No.300–330、No.370–390等区域的残基。笔者通过分析CSCE酶结晶结构发现,不同来源CE酶的活性口袋相似度很高,其中的氨基酸残基相当保守且有一定的疏水性,针对这些残基的定点突变实验都导致了酶活力大幅下降或彻底失活(结果尚未发表),这些残基的编号和推测功能如表4所述。Ito等分析了Ruminococcus albus来源CE酶(以依匹乳糖为产物)的重要氨基酸残基,结果表明无论底物是纤维二糖还是乳糖,R52、H243、E246、W249、W304、E308和H374残基都是催化必需残基,F114和W303也对催化有重要贡献[57]。Park等分析CSCE酶活性中心与甘露糖基团C-2位的作用情况,选择Y114和N184两个残基位点进行饱和突变研究。结果表明在所得突变体中,Y114E催化200 g/L乳糖生产乳果糖的转化率为43.5%(产物浓度86.9 g/L),尽管转化率不高但依匹乳糖的浓度仅为4.6 g/L (转化率2.3%),这可能是因为突变体丧失了大部分的差向异构化能力[36]。针对热稳定性的突变研究也有报道,Shen等发现双位点突变E161D/N365P在高温下的活性半衰期提高了4倍,最适反应温度也从80°C提高到87.5°C,对底物乳糖的催化效率kcat/Km上升了29%[58]。
| 图表编号 | XD00212138200 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2021.02.04 |
| 作者 | 徐铮、张倩、李克文、徐虹 |
| 绘制单位 | 南京工业大学食品与轻工学院材料化学工程国家重点实验室、保龄宝生物股份有限公司、保龄宝生物股份有限公司、南京工业大学食品与轻工学院材料化学工程国家重点实验室 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式