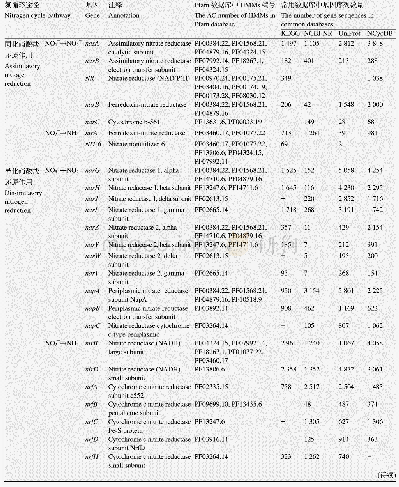
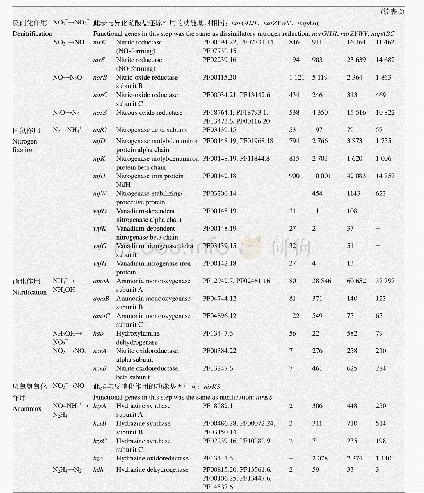
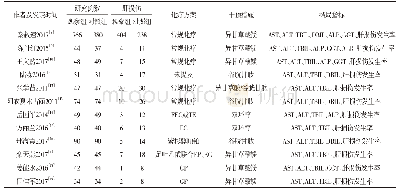
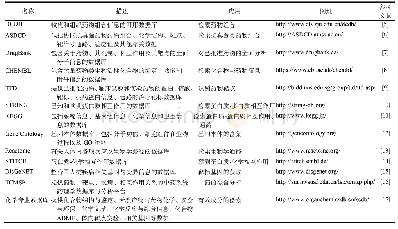
《表1 药物综合信息及其协同性相关的常用数据库》
基于药物信息学技术进行药物的协同性研究需要高质量数据的支持,充分利用已有的各类信息资源是重要的前提,一些常用的数据库见表1,如DCDB[5]、ASDCD[6]以及一些其他相关联的数据库[5-17],如Drug Bank[7]、CHEMBL[8]、TTD[9]、STRING[10]、KEGG[11]、Gene Ontology[12]等。其中,DCDB是收集和组织药物组合的数据库,该数据库根据>14万例的临床数据与FDA批准的橙皮书制定了包括1 363种药物组成的866种药物组合。ASDCD是抗真菌协同药物组合数据库,该数据库包括210种抗真菌增效药物组合和1 225种药物-靶标相互作用数据。Drug Bank是一个关于药物、机制、相互作用和目标的全面的分子信息数据库,包括2 358个FDA批准的药物。CHEMBL为几乎所有可用的小化合物提供化学信息,包括大量药物类生物活性化合物的绑定、功能和附件信息。TTD是一个专门用于注释已知和探索的大分子药物靶点的数据库,包括已批准药物、临床试验和实验药物的蛋白、核酸、靶标、疾病蛋白信息、通路的治疗靶标。KEGG为药物提供代谢信息,为药物靶点提供蛋白质注释,包括系统信息、基因组信息、化学信息和卫生信息。Gene Ontology(GO)是基因本体数据库,包括分子功能、细胞组件和生物过程3个方面,常用的软件有DAVID[18]、Cytoscape[19]、Metascape[20]等。
| 图表编号 | XD00211297000 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.12.28 |
| 作者 | 陈倩倩、李雪、陈石秀、鹿振辉、郑培永、杨铭 |
| 绘制单位 | 上海中医药大学附属龙华医院、上海中医药大学附属龙华医院、上海中医药大学附属龙华医院、上海中医药大学附属龙华医院、上海中医药大学附属龙华医院、上海中医药大学附属龙华医院 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式