《表2 基于全基因组测序数据的SSR位点挖掘》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《四倍体马铃薯抗卷叶病毒的分子标记开发及性状关联分析》
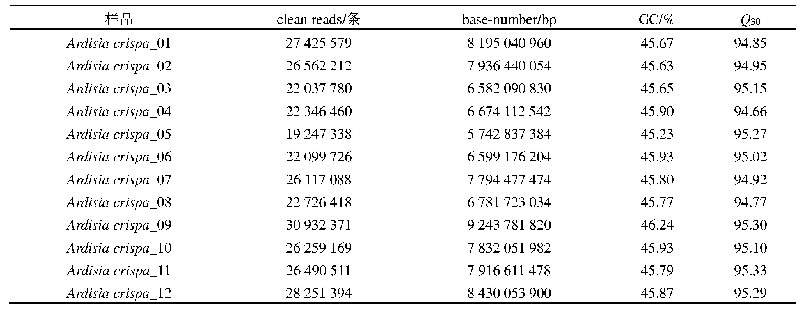
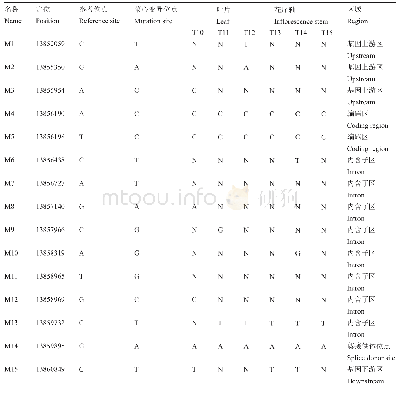
采用Mapping后的单倍体全基因数据,利用基于Perl语言的MISA软件进行SSR位点进行挖掘,结果发现在抗病群体60、65、140、185、189中挖掘到的SSR数目分别为17 550、20 693、20 068、18 648、19 919;在感病群体141、187、487、529、831中挖掘到的SSR数目分别为15 828、18 604、12 419、17 920、12 541。对抗病群体的5个单株进行组内过滤,筛选出542个共有SSR位点;对感病群体的5个单株进行组内比较,筛选出432个共有SSR位点。最后通过组间比较,筛选出277个特异性SSR位点,其中和抗病连锁的SSR位点有193个,感病连锁的SSR位点有84个(表2)。
| 图表编号 | XD00152876200 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.05.14 |
| 作者 | 杨祝强、梁静思、段晓艳、李灿辉、齐凤娜、谷爱新、杨洋、唐唯 |
| 绘制单位 | 云南师范大学生命科学学院、云南师范大学马铃薯科学研究院云南省马铃薯生物学重点实验室、云南师范大学生命科学学院、云南师范大学马铃薯科学研究院云南省马铃薯生物学重点实验室、云南师范大学生命科学学院、云南师范大学马铃薯科学研究院云南省马铃薯生物学重点实验室、云南师范大学生命科学学院、云南师范大学马铃薯科学研究院云南省马铃薯生物学重点实验室、云南师范大学生命科学学院、云南师范大学生命科学学院、云南师范大学生命科学学院、云南师范大学生命科学学院、云南师范大学马铃薯科学研究院云南省马铃薯生物学重点实验室 |
| 更多格式 | 高清、无水印(增值服务) |