《表1 经分子对接筛选得到的化合物结构信息表》
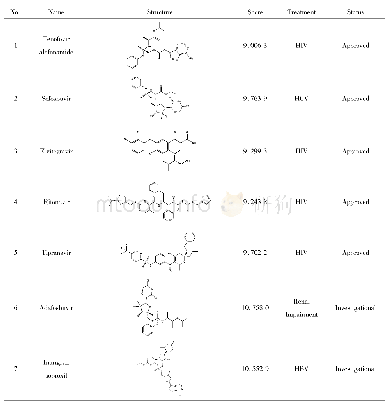
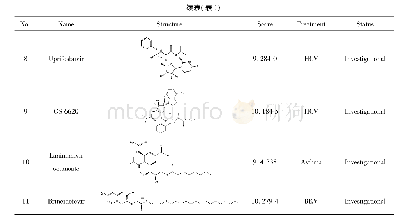
首先以阳性化合物pitstop2作为标准参照,评价对接方法的准确性所示(图1),通过分子对接获得的优势构象与阳性化合物原始构象的均方根偏差(root mean square difference,RMSD)为0.49?,说明两分子间叠合性较好。另外在对接所预测的pitstop2-4G55相互作用中,与配体分子产生紧密联系的氨基酸分别为Arg64、Ile66、Ile93、Ile52及Phe91等残基,其中Arg64提供氢键相互作用;Ile66、Ile93及Ile52等残基提供疏水相互作用;Phe91提供静电相互作用。这种相互作用模式与文献[21-24]所描述的“pitstop2结构中的噻唑酮环与受体蛋白的Arg64形成2个活性必需氢键,所引起的构象变化使得pitstop2的非极性部分深入由Ile52、Ile66和Ile62等残基组成的疏水空腔;此外,pitstop2的溴代苯环部分与受体蛋白的Phe91产生堆叠平面,形成阳离子-π相互作用”基本一致。表明本研究所运用的分子对接方法具有一定的可信度。紧接着对自有库中的化合物进行分子对接,预测各化合物与网格蛋白的结合位点并详细记录打分情况,最终共筛选出20个优选分子(表1和图2)。
| 图表编号 | XD00147539100 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.05.30 |
| 作者 | 李雨婉、李月香、曹瑞源、肖军海 |
| 绘制单位 | 北京军事科学院军事医学研究院毒物药物研究所、北京军事科学院军事医学研究院毒物药物研究所、北京军事科学院军事医学研究院毒物药物研究所、北京军事科学院军事医学研究院毒物药物研究所 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式