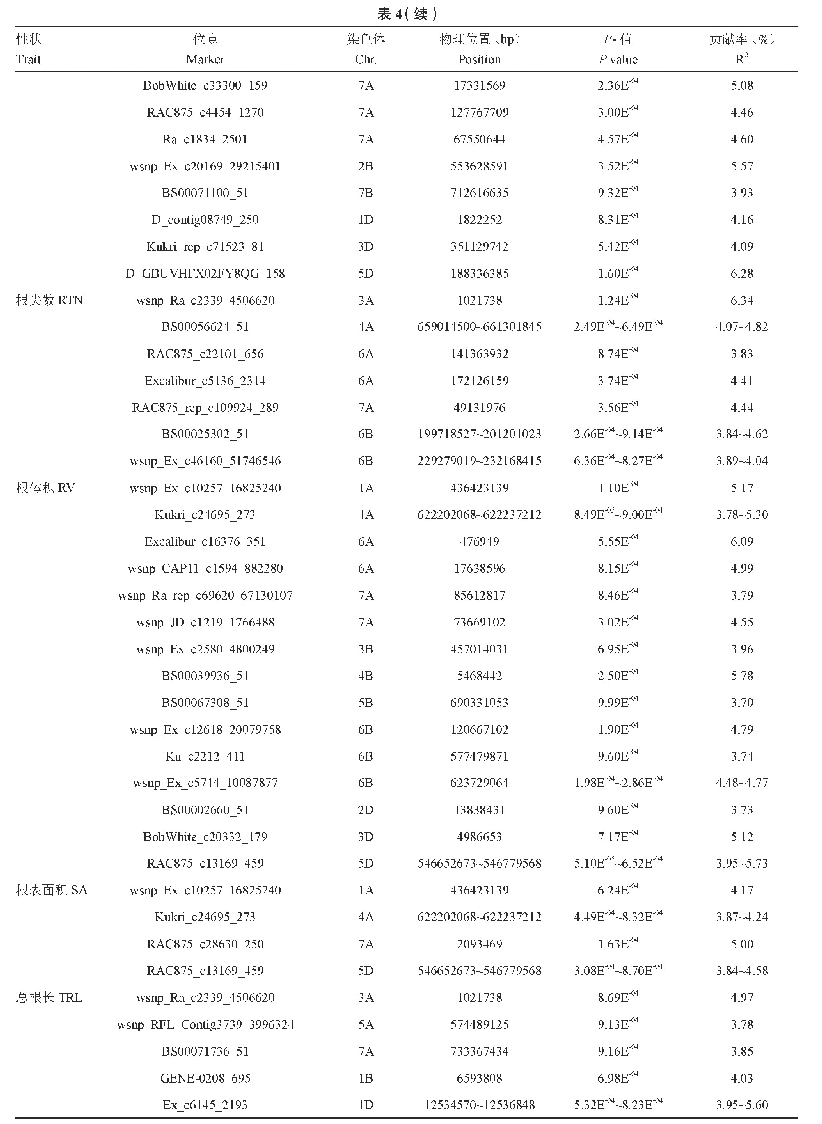
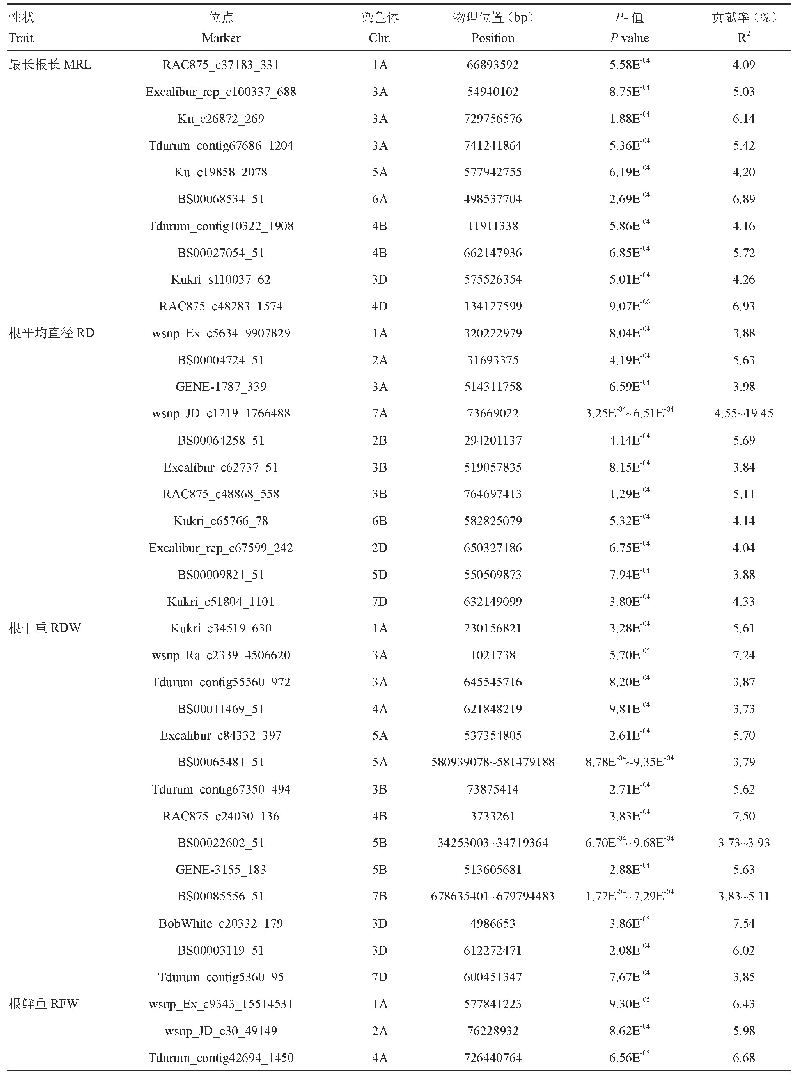
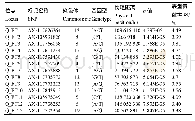
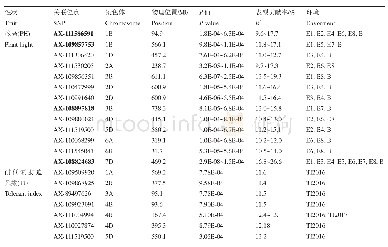
《表3 耐低磷相关性状显著关联的位点信息》
将198份试验材料耐低磷相关性状的耐低磷系数与11896个SNP标记进行全基因组关联分析,共检测到67个显著关联的标记(表3)。其中,检测到4个与单株苗茎叶部干重关联的SNP,分布在5D、6D染色体上,这些位点可解释的表型变异率分别为5.921%、5.826%、8.862%、6.654%。检测到11个与单株苗根部干重显著关联的SNP,分布在2D、6A、6B、6D染色体上,SNP可解释的表型变异率为5.859%~7.087%。与单株茎叶部磷累积量显著关联的SNP标记共检测到8个,分布在2A、6A、6B、6D、7B、7D染色体上,可解释的表型变异率为6.575%~9.097%。检测到38个与单株根部磷累积量显著关联的SNP标记,分布在1A、1B、1D、2A、2D、4A、4B、4D、5A、7A、7D、UN染色体上,标记位点可解释的表型变异率为5.892%~8.508%。检测到6个与单株根部磷利用效率关联的SNP,分布在2B、5A、5B、6A、7B染色体上,表型变异率为5.952%~9.552%。在这些显著关联的标记中,单株根部磷累积量关联到的标记最多(38个),且变异系数最大(26.59%),从一定程度上说明表型变异的大小会影响关联分析的关联效率。
| 图表编号 | XD00144033900 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.03.16 |
| 作者 | 周思远、毕惠惠、程西永、张旭睿、闰永行、王航辉、毛培钧、李海霞、许海霞 |
| 绘制单位 | 河南农业大学、省部共建小麦玉米作物学国家重点实验室、河南省粮食作物协同创新中心、河南农业大学、省部共建小麦玉米作物学国家重点实验室、河南省粮食作物协同创新中心、河南农业大学、省部共建小麦玉米作物学国家重点实验室、河南省粮食作物协同创新中心、河南农业大学、省部共建小麦玉米作物学国家重点实验室、河南省粮食作物协同创新中心、河南农业大学、省部共建小麦玉米作物学国家重点实验室、河南省粮食作物协同创新中心、河南农业大学、省部共建小麦玉米作物学国家重点实验室、河南省粮食作物协同创新中心、河南农业大学、省部共建小麦玉米 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式