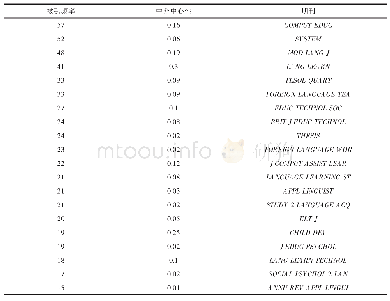
《表3 排名前20的microRNA与其修正P值》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《数据挖掘结合实验证明hsa-miR-371a-3p促进肝癌细胞迁移》
数据集分析整合以及分析结果验证的流程见图1(b).每个数据集分别进行标准化和分析,提取出上调和下调差异表达的microRNA列表,然后分别根据|logFC|从大到小进行排序.为了整合5个数据集的分析结果,我们引入了RobustRankAggregation(RRA)算法[18].该算法假设存在一个由所有差异表达的microRNA组成的池,然后计算每个microRNA在5个排序列表中随机分布的P值.P值越小的microRNA就越不可能是随机分布的,越有可能是真正差异表达的.同时,为了增加结果的稳健性,我们引入了交叉留一法(LOOCV)进行修正.具体方法是:随机舍弃5个数据集中的一个,对剩下的4个数据集使用RRA算法,记录每个microRNA的P值.重复运行10 000次.对每个microRNA的10 000个P值取中位数,作为这个microRNA的修正P值.针对上调和下调差异表达的microRNA列表分别进行分析.microRNA按照修正P值从小到大进行排序,保留排名前20的microRNA进行后续分析.排名前20的microRNA与其修正P值见表3.
| 图表编号 | XD00130850500 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.04.01 |
| 作者 | 何懿、吴家雪 |
| 绘制单位 | 复旦大学生命科学学院、复旦大学生命科学学院 |
| 更多格式 | 高清、无水印(增值服务) |