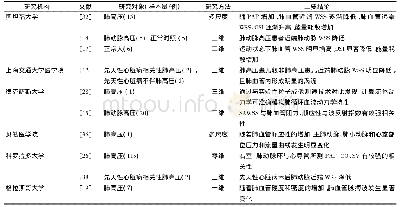
《表1 近几年全基因组关联分析在细菌中应用的文献》
GWAS是基因组分析的重要工具,并且近几年已经成功应用到细菌基因组分析上(表1)。早期的细菌基因组主要基于PCR技术或比较基因组杂交技术,通过与表型结合,以寻找基因组中特定基因的存在或缺失与表型的关系,然而因为成本和平台的限制,以及某些细菌自身的核苷酸多样性较低,如炭疽杆菌(Bacillus anthracis)[28],使得其无法在细菌基因组中大规模应用。2013年Sheppard等[29]首次实现GWAS在细菌中的应用,将192株分离自牛和鸡肠道的弯曲杆菌(Campy鄄lobacter)的DNA序列划分成30 bp长的“word”来寻找菌株与特定宿主定殖能力方面的相关性,共检测到7 307个“word”与弯曲杆菌在牛肠道中的特异性定殖有关,注释到3个与维生素B5生物合成相关基因,因而认为弯曲杆菌自身能否合成维生素B5是其能否在牛肠道定殖的关键。之后Chewapreecha等[30]的研究首次使GWAS在细菌中的研究达到了人类遗传学研究的规模,他们总共分析了收集自缅甸和泰国边境的3 085株和收集自美国马萨诸塞州的616株肺炎链球菌(Strepto鄄coccus pneumoniae)用于检测β-内酰胺抗性相关的基因组变异。
| 图表编号 | XD00113997200 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.10.31 |
| 作者 | 余中节、赵洁、宋宇琴、孙志宏、张和平 |
| 绘制单位 | 内蒙古农业大学乳品生物技术与工程教育部重点实验室农业部奶制品加工重点实验室内蒙古自治区乳品生物技术与工程重点实验室、内蒙古农业大学乳品生物技术与工程教育部重点实验室农业部奶制品加工重点实验室内蒙古自治区乳品生物技术与工程重点实验室、内蒙古农业大学乳品生物技术与工程教育部重点实验室农业部奶制品加工重点实验室内蒙古自治区乳品生物技术与工程重点实验室、内蒙古农业大学乳品生物技术与工程教育部重点实验室农业部奶制品加工重点实验室内蒙古自治区乳品生物技术与工程重点实验室、内蒙古农业大学乳品生物技术与工程教育部重点实验 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式