《表1 SNP分布情况:蒙古羊不同尾椎数相关选择信号的挖掘研究》
注:upstream:基因上游1 kb区域;exonic:变异位于外显子区域;intronic:变异位于内含子区域;splicing:变异位于剪接位点(内含子中靠近外显子/内含子边界的2bp);downstream:基因下游1 kb区域;intergenic:变异位于基因间区。
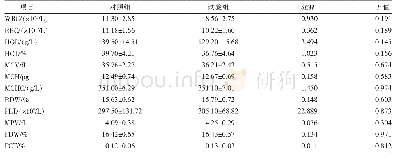
该研究对28只蒙古羊进行全基因组重测序,样本平均有效测序深度达到6×(绵羊基因组大小为2.6 Gb),测序质量较高(Q20=96.31%,Q30=91.71%),GC分布正常,GC含量为44.38%,对基因组的覆盖度均大于99%。通过GATK和samtools软件共同分析得到可靠SNP数量为29 926 218,Indels数量为3 755 604。SNP的分布情况见表1,其主要分布在基因间区和内含子区。
| 图表编号 | XD0038586500 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.01.30 |
| 作者 | 达赖、付绍印、刘斌、何小龙、王标、陈欣、方勤圆、张兴夫、姜卓、张文广、刘永斌 |
| 绘制单位 | 内蒙古自治区农牧业科学院、内蒙古自治区农牧业科学院、内蒙古ATCG生物信息研究所、内蒙古犇牛科技有限公司、内蒙古自治区农牧业科学院、内蒙古自治区农牧业科学院、内蒙古自治区农牧业科学院、内蒙古自治区农牧业科学院、内蒙古自治区农牧业科学院、扎兰屯市农牧业局畜牧工作站、内蒙古ATCG生物信息研究所、内蒙古犇牛科技有限公司、内蒙古农业大学动物科学学院、内蒙古自治区农牧业科学院 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式