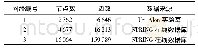《表2 生物网络数据集Tab.2 Biological network datasets》
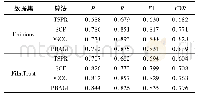
实验同时使用了模拟数据和真实生物网络数据进行性能分析,其中模拟数据使用Network X[13]的Python包模拟了三类不同的网络模型,分别是无标度网络模型[14]、小世界网络模型[15]和规则网络模型[16],每类网络模型随机地改变边数和节点数,产生具有不同平均节点度的网络,以探讨在不同的平均节点度下并行ESU算法的性能。实验中使用固定的4 000个节点,同时变化边的条数,分别为20000、40000和60000条边,来产生不同的节点平均度,分别是5、10和15,每种平均节点度模型均生成10个网络;真实生物网络则选取了酵母菌代谢网络、酵母菌蛋白质网络和人类蛋白质网络三个生物网络数据集,其节点数和边数如表2所示,其中:酵母菌的蛋白质网络和代谢网络数据集来源于Uri Alon实验室[2],人类蛋白质网络数据来源于STRING(Search Tool for Recurring Instances of Neighbouring Genes)在线数据库[17]。
| 图表编号 | XD0035543900 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.01.10 |
| 作者 | 杨伏长、朱嘉富、孙佳敏、谢江 |
| 绘制单位 | 上海大学计算机工程与科学学院、上海大学计算机工程与科学学院、上海大学计算机工程与科学学院、上海大学计算机工程与科学学院 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式