《表3 不同模型在JNLPBA数据集上的评价指标对比》
单位:%
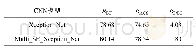
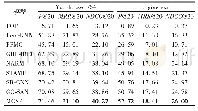
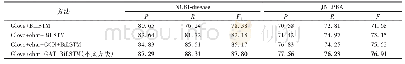
各模型在JNLPBA数据集上的表现对比如表3所示。Tang等[20]在生物医学命名实体识别中融入词表示特征;Li等[21]提出在模型中结合使用句子向量和双词向量;Wei等[22]在Bi LSTM-CRF模型中引入注意力机制,获得了71.57%的准确率和73.50%的F1值;Dai等[23]在Bi LSTM-CRF模型中使用预训练词向量模型ELMO(Embeddings from Language MOdels,ELMO),获得了74.29%的F1值。以上方法均没有利用语料中的句法信息,效果有所局限。本文模型使用CNN提取字符特征,帮助模型识别包含特殊字符的实体,在不使用人工设计特征的情况下,提升了实体识别率;其次,本文使用GCN学习语料中的句法依存分析信息,增强了词与词之间的关联。GCN在归一化操作后会为每个节点的邻接节点赋予相同的权重,忽视了节点之间依存关系的差异性。针对这一问题,本文还引入了图注意力机制优化邻接节点的特征权重,让模型更好地聚合邻接节点特征。实验结果表明,本文模型的F1值比Dai等[23]提出的模型高出了2.62个百分点,相比于Wei等[22]提出的模型,其准确率提高了5.99个百分点,F1值提高了3.41个百分点,获得了更好表现。
| 图表编号 | XD00201792500 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2021.02.10 |
| 作者 | 许力、李建华 |
| 绘制单位 | 华东理工大学信息科学与工程学院、华东理工大学信息科学与工程学院 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式