《表4 不同模型在NCBI数据集上的评价指标对比》
单位:%
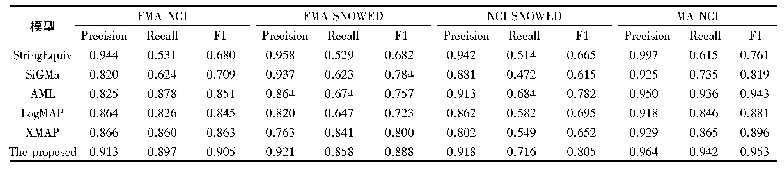
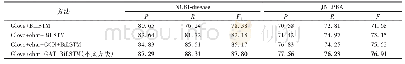
模型在NCBI-disease数据集上的表现对比如表4所示。Leaman等[24]提出的DNorm模型使用CRF结合多种人工设计特征;Lu等[25]提出的Tagger One模型通过正则化方法降低实体识别错误率;Dang等[8]提出的D3NER模型在Bi LSTM-CRF模型中引入语言学特征对词向量进行优化,取得了85.03%的准确率和84.41%的F1值;Wang等[26]在Bi LSTM-CRF模型中融入多任务学习思想,同时学习多种生物医学实体类型,取得了85.86%的准确率和86.14%的F1值。以上这些方法的思路大多是通过融合特征或优化词向量来提升模型效果,没有从句法的角度考虑词与词之间的关联。本文模型利用GCN学习文本的句法依存信息,并引入图注意力机制优化邻接节点的特征权重,充分地利用了语料中的句法信息。实验结果表明,相比Wang等[26]提出的模型,本文模型的准确率提升了1.43个百分点,F1值提升了1.66个百分点;相比D3NER模型[8],准确率提升了2.26个百分点,F1值提升了3.39个百分点,取得了良好的效果。
| 图表编号 | XD00201792100 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2021.02.10 |
| 作者 | 许力、李建华 |
| 绘制单位 | 华东理工大学信息科学与工程学院、华东理工大学信息科学与工程学院 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式