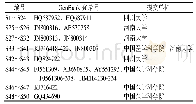
《表2 柴胡序列相关信息:基于ITS2条形码鉴定藏柴胡及其易混品》
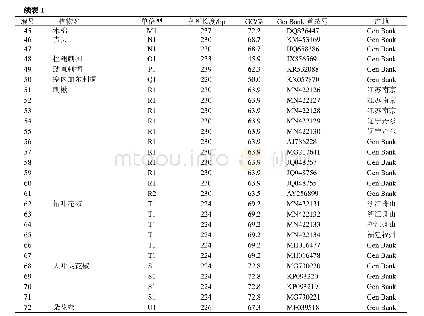
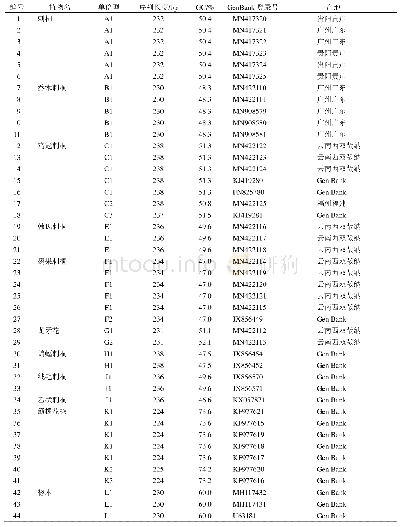
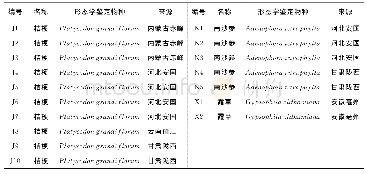
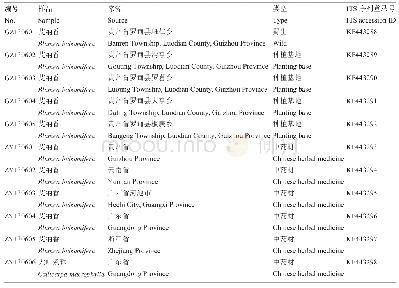
应用Codon Code Aligner软件对测序峰图进行校对拼接,基于隐马尔可夫模型的HMMer注释方法[36],手动查找并去除两端5.8 S和28 S区段(图1),获得ITS2间隔区序列。登陆Gen Bank的Nucleotide数据库(http://www.ncbi.nlm.nih.gov/nuccore)下载相关柴胡序列(下载信息见表2)。利用MEGA(molecular evolutionary genetics analysis)6.0进行序列比对,计算GC含量及种内、种间K2P(Kimura 2-parameter)遗传距离,采用邻接法(neighbour-joining,NJ)构建系统聚类树,同时以bootstrap(自展支持率1 000次)重复检验各分支的支持率。根据Koetschan等[37-38]建立的ITS2数据库及其网站预测ITS2的二级结构。
| 图表编号 | XD00200711600 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.12.12 |
| 作者 | 夏召弟、刘霞、冯玛莉、席啸虎、仝立国 |
| 绘制单位 | 山西省中医药研究院、山西省中医药研究院、山西省中医药研究院、山西省中医药研究院、山西省中医药研究院 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式