《表2 基于大豆地方品种资源群体的全基因组关联分析方法比较》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《限制性两阶段多位点全基因组关联分析法在遗传育种中的应用》
h2:性状遗传率估计;R2:QTL表型变异解释率;MLM:混合线性模型方法
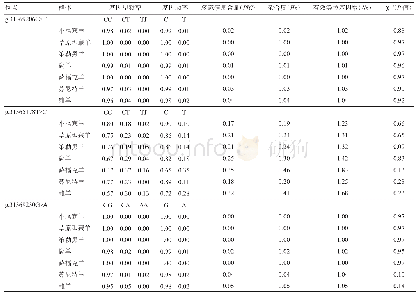
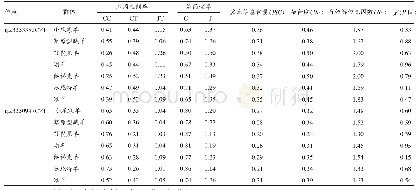
自然群体遗传变异丰富,作物种质资源群体更是品种改良的重要基因资源。全面解析自然群体/资源群体大量存在的QTL及其复等位变异将有助于了解数量性状的遗传规律以及植物遗传改良。ZHANG等[21]基于由366份地方大豆材料组成的资源群体,分别使用了RTM-GWAS方法和目前最常用的MLM方法对油脂、油酸和亚麻酸含量进行GWAS分析(表2)。结果显示,在Bonferroni多重测验矫正下,MLM方法对3个性状分别检测到3、18和22个QTL,表型变异解释率分别是19.69%、138.76%和206.52%。可见MLM方法检测的QTL不仅偏少,而且油酸和亚麻酸含量QTL的表型变异解释率还远超过性状遗传率,表明MLM方法中QTL效应估计偏差较大。而RTM-GWAS方法分别检测到50、98和50个QTL,表型变异解释率分别是82.53%、90.29%和83.84%,均小于性状遗传率,结果更为合理。
| 图表编号 | XD00180888100 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.05.01 |
| 作者 | 贺建波、刘方东、王吴彬、邢光南、管荣展、盖钧镒 |
| 绘制单位 | 南京农业大学大豆研究所、国家大豆改良中心、农业部大豆生物学与遗传育种重点实验室、作物遗传与种质创新国家重点实验室、江苏省现代作物生产协同创新中心、南京农业大学大豆研究所、国家大豆改良中心、农业部大豆生物学与遗传育种重点实验室、作物遗传与种质创新国家重点实验室、江苏省现代作物生产协同创新中心、南京农业大学大豆研究所、国家大豆改良中心、农业部大豆生物学与遗传育种重点实验室、作物遗传与种质创新国家重点实验室、江苏省现代作物生产协同创新中心、南京农业大学大豆研究所、国家大豆改良中心、农业部大豆生物学与遗传育种重点 |
| 更多格式 | 高清、无水印(增值服务) |