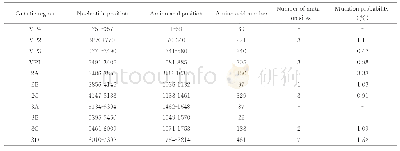
《表2 氨基酸虚拟突变后结构稳定的突变体》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《"定向引入N-糖基化位点改进黑曲霉β-1,4-内切木聚糖酶热稳定性"》
注:Stabilizing表示突变能<-0.5 kcal/mol;Neutral表示-0.5 kcal/mol≤突变能≤0.5 kcal/mol.
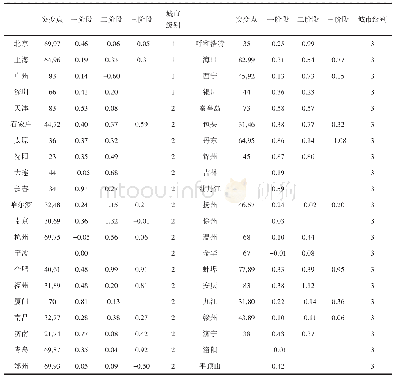
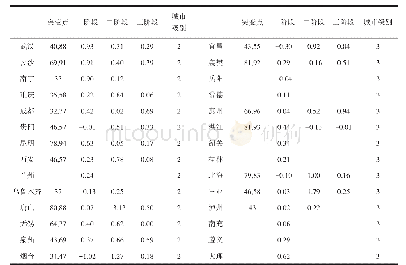
(1)利用DS 4.5对xynB模型进行RSA分析,表征暴露在蛋白表面的氨基酸残基(RSA>25%)的分布(图2A);(2)记录暴露在xynB表面β-转角处和Loop区可构建N-糖基化的氨基酸序列作为可突变序列,共有13条(表1);(3)xynB模型与底物木五糖的CDOCKER对接结果显示xynB与底物结合时结合面氨基酸有Ser52,Asn80,Tyr108、206、112、123、214,Arg157,Ala161,Trp54、114、173,Val82,Phe81,Gln171和Glu212(图2B);(4)结合面氨基酸经丙氨酸扫描获得热点残基为Tyr112、206,Trp173,Gln171和Arg157,舍弃含有热点残基的可突变序列;(5)同时舍弃突变后序列Asn-X-Ser/Thr-Y中Y位置为Pro、Glu和Trp的可突变序列;(6)根据N-X-S/T、F-Y-Z-N-X-S/T、F-N-X-S/T、F-X-N-X-S/T序列对候选可突变序列进行氨基酸虚拟突变,保留突变能≤0.5 kcal/mol的突变体。经以上6步筛选得到10个结构稳定的突变体(表2)。
| 图表编号 | XD00174031800 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.07.20 |
| 作者 | 吴凤梅、周燕霞、姚冬生、谢春芳 |
| 绘制单位 | 暨南大学生物医药研究院、暨南大学基因工程药物国家工程研究中心、暨南大学生物医药研究院、暨南大学基因工程药物国家工程研究中心、暨南大学生物医药研究院、暨南大学基因工程药物国家工程研究中心、暨南大学生命科学技术学院生物工程学系 |
| 更多格式 | 高清、无水印(增值服务) |