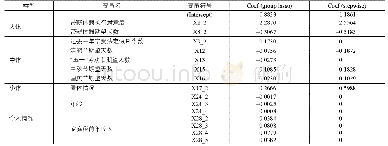
《表1 用Lasso回归和岭回归对水稻4个性状进行全基因组预测的效果》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《基于JMP软件的Lasso及岭回归在水稻全基因组预测中的应用》
**表示相关系数在α=0.01的水平上具有统计学意义
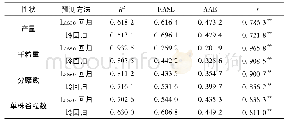
表1列出了用岭回归和Lasso回归对产量、千粒重、分蘖数及单株谷粒数等4个性状进行全基因组预测的模型性能和预测效果评价指标的估计值,并在图1中对两种预测方法和不同性状的预测效果进行了比较.决定系数(R2)反映的是模型的拟合优度,均方根误差RASE和平均绝对误差AAE也是模型性能评价的常用指标,其中AAE受离群值影响较小;预测值与实际值的相关系数(r)在全基因组选择中通常被用来衡量预测的准确性.表1和图1的结果表明,两种预测方法对于4个性状都有较好的预测效果(最小的r=0.721 8),但Lasso回归的模型拟合及预测效果一致地优于岭回归,其中拟合最好的是千粒重的Lasso回归预测模型(R2=0.932 5),即模型解释了该性状变异的93.25%.图2是各性状的实际值-预测值图,从中可以看出岭回归预测值的变异性都大于Lasso回归.对于这两种预测方法,4个性状的模型拟合及预测效果的次序为:千粒重>分蘖数>单株谷粒数>产量.
| 图表编号 | XD00158689900 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.07.05 |
| 作者 | 李亚男、陈建国 |
| 绘制单位 | 湖北大学生命科学学院、湖北大学生命科学学院 |
| 更多格式 | 高清、无水印(增值服务) |