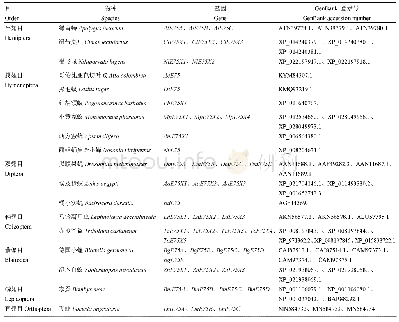
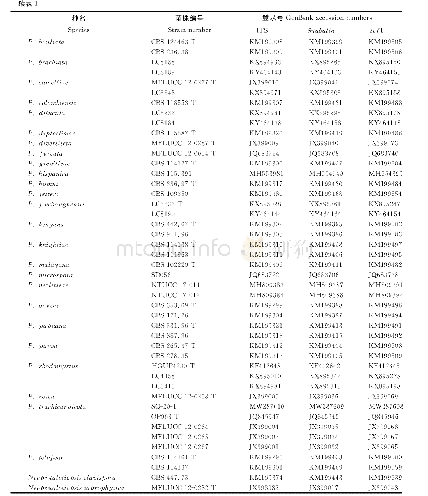
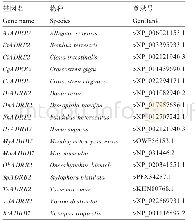
《表4 基于UPGMA、NJ法构建进化树的平均节点支持率和多序列同质性检验》
注:1)括号中数值-节点支持率≥70的节点数百分比
基于UPGMA和NJ法两种方法,分析单一片段和多序列片段组合构建进化树平均节点支持率的情况(表4)。其中无论是UP-GMA还是NJ法,单一片段构建进化树的平均节点支持率都比多序列片段组合构建进化树的平均节点支持率低。两种方法构建进化树平均节点支持率较高的组合均为mat K+ITS2、mat K+rbc L、mat K+ITS2+rbc L、mat K+ITS2+trnH-psbA、matK+rbcL+trnH-psbA和matK+ITS2+rbcL+trnH-psbA,同质性检验也表明这些组合构建进化树均呈现显著关系(P<0.05),并适用于进化树构建。其中,mat K+ITS2+rbcL和matK+ITS2+rbcL+trnH-psbA建树效果最佳,matK+ITS2+rbc L基于UPGMA法构建进化树的平均节点支持率为58.60(53.33%),NJ法构建进化树的平均节点支持率为56.47(53.33%);同理可知,mat K+ITS2+rbc L+trn H-psbA组合UPG-MA法为59.67(53.33%),NJ法为54.27(53.33%)。综合比较而言,最终选择mat K+ITS2+rbc L组合构建多个沉香属物种进化树。
| 图表编号 | XD00128049600 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.12.08 |
| 作者 | 康勇、刘杨、杨云、冯剑、郑希龙、魏建和 |
| 绘制单位 | 中国医学科学院北京协和医院药用植物研究所海南分所海南省南药资源保护与开发重点实验室国家中医药管理局沉香可持续利用重点研究室、中国医学科学院北京协和医院药用植物研究所中草药物质基础与资源利用教育部重点实验室濒危药材繁育国家工程实验室、中国医学科学院北京协和医院药用植物研究所海南分所海南省南药资源保护与开发重点实验室国家中医药管理局沉香可持续利用重点研究室、中国医学科学院北京协和医院药用植物研究所海南分所海南省南药资源保护与开发重点实验室国家中医药管理局沉香可持续利用重点研究室、中国医学科学院北京协和医院药用 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式