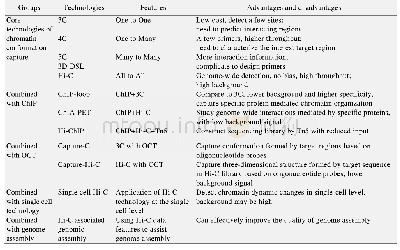
《表2 4种目标序列捕获技术的比较》
目标序列捕获技术是基于DNA杂交原理,通过专门设计的探针与基因组序列进行杂交,富集基因组中特定区域并进行高通量测序的方法.该方法的一般流程是,首先通过参考基因组序列比对分析选择可以富集的保守序列区域,根据序列的保守性设计合成具有生物素标记的长度为80~120 bp的DNA或RNA单链探针,随后将探针与目标样本的基因组测序文库进行杂交并固定在生物素亲合磁珠上,最后洗脱未杂交上的文库片段,回收目标文库片段进行高通量测序[58].序列捕获技术对DNA质量要求较低,适用于DNA降解较为严重的样本[59,60],同时实验操作技术和设计捕获探针的生物信息学流程较为成熟,在捕获目标区域的长度和捕获基因的数量上有较高的灵活性,数据回收率和利用率较高,因而近年来已经成为了系统发育基因组学获取数据的主流方法.目前常用的目标序列捕获方案和特点如表2所示.其中锚定序列捕获(anchored enrichment,AHE)[61]和超保守序列元件捕获技术(ultraconserved element,UCE)[62]仅通过一套通用探针就能够对进化跨度大的不同物种进行序列捕获[69~73].如Prum等人[74]应用AHE技术对198个鸟类物种进行序列捕获,成功构建了高解析度的鸟类的系统发育关系.目前商业化探针的主要供应商有Aglient SureSelect,Roche NimbleGen和MyBait等,价格都比较昂贵,因此,为了降低实验成本,许多学者将线粒体基因组或基因组目标区域的PCR扩增产物作为捕获探针,进行目标序列捕获,也得到了不错的效果[66~68].这类实验室自制探针的方法,实验成本低、灵活性强,可应用在不同生物类群当中.然而,由于基因片段数量的限制,自制探针的方法能捕获到的标记数目一般较少,而且由于分子标记引物设计需要相关的参考基因组序列,对非模式生物类群的应用会受到一定的限制[75].
| 图表编号 | XD0034731500 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.04.20 |
| 作者 | 李佳璇、梁丹、张鹏 |
| 绘制单位 | 中山大学生命科学学院、中山大学生命科学学院、中山大学生命科学学院 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式