《表4 Salinas数据集的纹理特征相关矩阵》
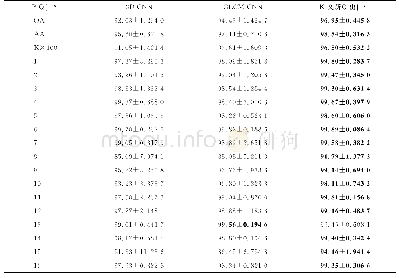
CNN中包含大量待训练参数,而网络的结构直接影响训练参数的多少,结构越复杂,训练参数越多,同时需要训练出成熟稳定的模型所耗时间也就越多。本文通过GLCM算法从第一主成分波段提取8个纹理特征,其中滑动窗口的大小均为5×5。对于近邻未标签样本的空间信息,采用相关性分析的方法,去除纹理特征中相关性较大的成分。表4、表5、表6分别为Salinas、Indian、Pavia University3个数据集的纹理特征相关系数矩阵。在通过相关系数矩阵进行相关性分析时,有时需要去除冗余的变量成分。2个变量相关性较大时,可以对比变量与其他变量的相关性,保留整体相关性较小的变量。当2个纹理特征高度相关(相关系数绝对值大于0.8)时,则选择平均绝对相关性(average absolute correlation,AAC)[2]较小的纹理成分进一步研究。某一特征的AAC为该特征与其他特征(含该特征本身)的相关系数绝对值和的平均。例如,表4中,Hom(同质度)与Ent(熵)之间的相关系数为-0.931 1,Hom的平均绝对相关值为0.583 2,Ent的平均绝对相关值为0.561 9,则去除高相关成分Hom。因此最终Salinas数据集中保留的纹理成分为均值(Mea)、方差(Var)、对比度(Con)、相异性(Dis)、角二阶矩(ASM)、相关性(Cor)。Indian数据集保留的纹理成分为均值(Mea)、方差(Var)、熵(Ent)、角二阶矩(ASM)、相关性(Cor)。Pavia University数据集保留的纹理成分为均值(Mea)、对比度(Con)、角二阶矩(ASM)、相关性(Cor)。
| 图表编号 | XD00199354700 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.10.20 |
| 作者 | 韩彦岭、高仪、王静、张云、洪中华 |
| 绘制单位 | 上海海洋大学信息学院、上海海洋大学信息学院、上海海洋大学信息学院、上海海洋大学信息学院、上海海洋大学信息学院 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式