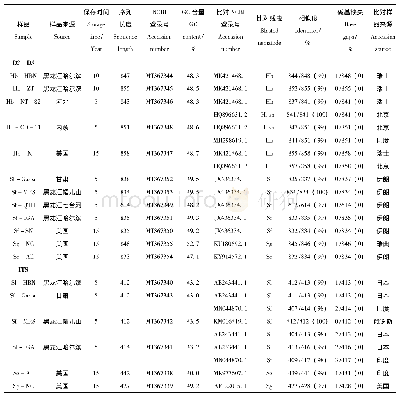
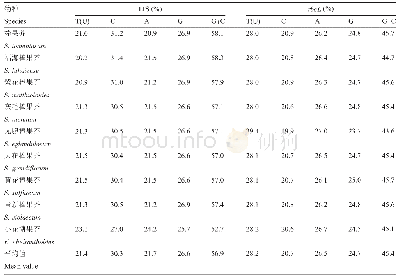
《表2 25个多花黄精样品的ITS2和psbA-trnH序列长度和碱基分布频率》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《基于DNA条形码的多花黄精系统发育和变异位点分析研究》
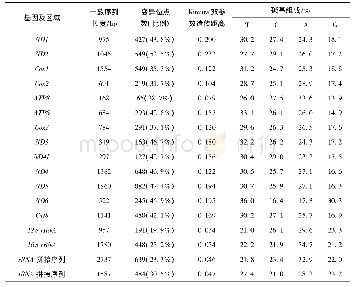
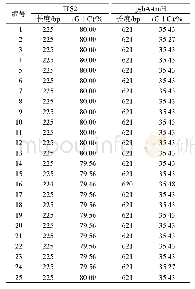
本研究分别扩增了25份多花黄精资源的ITS序列和psbA-trnH区段DNA序列。ITS2序列包括部分5.8 S rRNA序列+ITS2序列+部分28 S rRNA序列,psbA-trnH序列包括psbA基因全长+psbA-trnH间区全长+trnH基因全长。参照KX375075和KJ745884序列提取25个样品的ITS2和psbA-trnH序列后进行分析,安徽青阳样品的ITS2序列和psbA-trnH序列分别为224 bp和620bp,其余24个样品的ITS2序列和psb A-trn H序列分别为225 bp和621 bp。这些样品的ITS2序列和psbA-trnH序列在G+C含量方面较为接近(表2)。以滇黄精ITS2序列和psb A-trn H序列为外群,用邻接法基于ITS2序列和psb A-trn H序列构建系统发育树(图1)。结果显示,ITS2序列可将25份多花黄精资源分为2个大的类群,其中来自湖南洪江、新宁和资兴的样品和来自贵州花溪、剑河的样品组成一个类群,其中贵州剑河和花溪的样品又组成一个小的类群;其余20份资源组成另一个大的类群,其中湖北通山、雅安和麻城的样品组成一个小的类群,安徽青阳和浙江遂昌的样品组成一个小的类群,其余的来自福建、江西和广东的15个样品则无法区分开,基于ITS2序列构建的系统树显示出多花黄精资源具有地理遗传多样性。基于psb A-trn H序列构建的系统发育树则无法将25个样品区分开,但来自贵州剑河、福建泰宁和福建建阳的样品同其他22个样品具有一定差异。相对psbA-trnH序列,ITS2序列构建的系统发育树对不同地域来源的多花黄精样品区分度更好。
| 图表编号 | XD00178436900 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.08.12 |
| 作者 | 周先治、饶宝蓉、高晖、陈阳、林永胜 |
| 绘制单位 | 福建省农业科学院农业生物资源研究所、农业农村部福州热带作物科学观测实验站、福建省南平市农业科学研究所、福建省农业科学院农业生物资源研究所、福建省农业科学院农业生物资源研究所、福建省农业科学院农业生物资源研究所 |
| 更多格式 | 高清、无水印(增值服务) |