《表4 真实数据集上正确对准的百分比》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《通过区域筛选和简洁de Bruijn图比对高重复短序列》
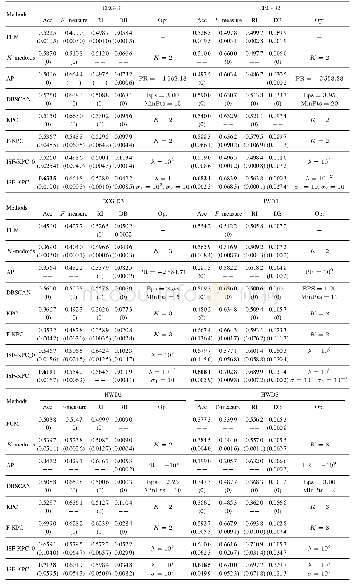
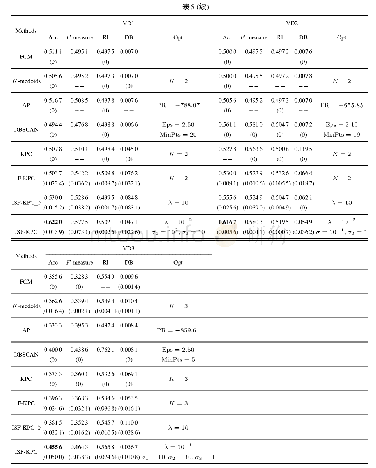
为评测算法在真实数据集上进行序列比对的效果,表4给出4种算法在8个真实数据集R1~R8上正确对准测序序列与参考基因组的百分比.表4的结果表明,本文算法SRA-RFCdBG在细菌基因组(R1-R5)和真核基因组(R7,R8)上正确对准序列与参考基因组的百分比均高于其他3种算法.这是因为算法SRA-RFCdBG采用基于空位种子的区域选择方法对种子进行筛选,根据DC碱基构造的不同类型的查找关键字构建hash索引,索引查找两个满足条件的互补shape,从而完成近似匹配的敏感查找,这样即使在空位中存在“indel”的情形,也可以找到对应匹配的种子;而其他3种算法未考虑该情形对比对结果的影响,未能找出空位中存在“indel”错误的匹配的种子,这导致了较低的正确比对百分比.对于人类基因组的真实数据集R6,算法SRA-RFCdBG的正确对准百分比略微比deBGA的正确对准百分比低0.10%,但高于其他2种算法BrownieAligner和BGREAT的正确对准百分比.整体而言,相较于其他3种算法,算法SRA-RFCdBG在8个数据集上获得了更高的正确对准百分比.
| 图表编号 | XD00157821200 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.09.01 |
| 作者 | 黎瑶、钟诚 |
| 绘制单位 | 广西大学计算机与电子信息学院广西高校并行分布式计算技术重点实验室、广西大学计算机与电子信息学院广西高校并行分布式计算技术重点实验室 |
| 更多格式 | 高清、无水印(增值服务) |