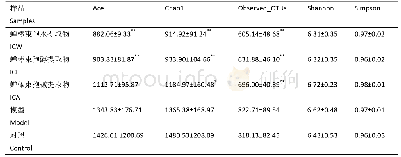
《表3 不同时间点放牧蒙古牛肠道菌群α多样性指数》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《内蒙古锡林郭勒冬季放牧蒙古牛肠道微生物多样性和功能预测分析》
基于加权的UniFrac距离进行了PCoA分析,并构建了系统发育UPGMA聚类树,分析样品间微生物的相似性。由图2可知,主成分PC1和PC2的贡献率分别为总变量的41.89%和16.55%,11和12月时的放牧蒙古牛样本相互聚集在一起,而次年1月时的蒙古牛样本则离散的较远,单独聚为一簇,说明寒冷的次年1月时的样本微生物群落组成与其他时间点差异性较大。基于加权的UniFrac距离计算后构建的系统发育树UPGMA聚类树得出了与PCoA分析相似的结论(图3),次年1月的样本与11和12月能够明显的区分开,11和12月的样品聚为一类,说明这两个时间点菌群组成有一定相似性。而与次年1月时具有差异性,这可能是由于次年1月时天气最为寒冷,牧草营养价值低,从而导致与其他时间点的蒙古牛样本的细菌多样性产生差异。
| 图表编号 | XD00152845200 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.10.01 |
| 作者 | 阿日查、斯木吉德、王纯洁、张剑、陈浩、刘波、何利娜、张晨、崔银雪、敖日格乐 |
| 绘制单位 | 内蒙古农业大学动物科学学院、内蒙古农业大学动物科学学院、内蒙古农业大学兽医学院、内蒙古农业大学动物科学学院、内蒙古农业大学动物科学学院、内蒙古农业大学动物科学学院、内蒙古农业大学动物科学学院、内蒙古农业大学动物科学学院、内蒙古农业大学动物科学学院、内蒙古农业大学动物科学学院 |
| 更多格式 | 高清、无水印(增值服务) |