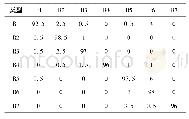
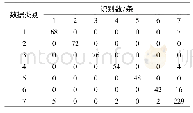
《表1 1 第三条DNA序列混淆矩阵对外显子识别结果》
如图3所示,数据框沿着测试DNA序列滑动,位于数据框结尾位置的碱基为C,数据框中包含有碱基C及其上游的一共250个碱基。如果被截取的序列被模型判定为外显子,则说明碱基C之前的250个碱基构成的序列被判定为外显子,此时模型的输出值为“1”。然后数据框将沿着DNA序列向前移动一个碱基,此时数据框结尾处的碱基为G,若此时模型的输出值为“1”则说明碱基G也为外显子,若模型的输出值为“2”则说明碱基G被模型判定为内含子中的碱基。以此类推,一条长度为N的DNA序列可以被切分成N-249条测试序列放入模型中进行判别,从而确定该条序列中外显子和内含子的具体位置和数量。本文所采用的5条测试序列长度分别为2 176、4 775、13 054、7 658、3 967。为了更清晰地展示本文所提出的模型对蛋白质编码区的预测效果,利用混合矩阵将预测值和真实值进行比较,结果如表9~表13所示,其中1表示外显子,2表示内含子。
| 图表编号 | XD00119818700 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.02.15 |
| 作者 | 胡青渝、刘广臣 |
| 绘制单位 | 鲁东大学数学与统计科学学院、重庆大学数学与统计学院、鲁东大学数学与统计科学学院 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式