《表1 mRNA中m6 A检测方法的比较》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《mRNA中N~6-甲基腺苷修饰及其在动物中的研究进展》
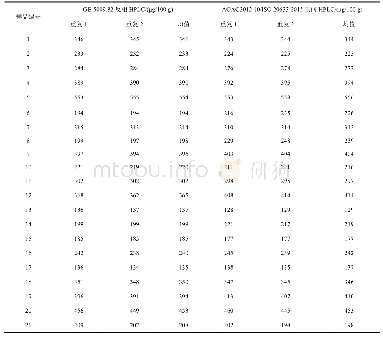
为了揭示m6 A的生物学功能,首先需要确定其在mRNA转录本中的位置。由于mRNA中m6 A修饰不影响Watson-Crick碱基配对原则,这使得基于逆转录的方法检测不到其存在,致使难度增加。其次,由于在转录组范围内缺乏检测表观转录组学标记的敏感测序技术,也使得表观转录组学研究落后于表观基因组学。虽然科学家们通过薄层层析(two-dimensional thin layer chromatography,2D-TLC)、斑点杂交(dot-blot)和液相色谱串联质谱(liquid chromatography-tandem mass spectrometry,LC-MS/MS)等一些传统方法也能检测到转录组中m6 A的分布,但是这些方法具有很大的局限性,在很大程度上依赖于物理化学技术,无法满足整个转录组水平上m6 A分布的研究要求。直到2012年,两个研究小组独立开发了高通量测序与抗体免疫沉淀相结合的方法(m6 A-Seq或MeRIP-Seq,methylated RNA immunoprecipitation and sequencing),这样才能够在整个转录组范围内检测m6 A的分布[1-2]。该方法是近年来mRNA修饰研究历程中的一大进步,但同样也存在一定的局限性,例如对原始材料的质量和数量要求高、受抗体影响大、分辨率低等。随后科学家们又开发了SCARLET(sitespecific cleavage and radioactive-labeling followed by ligation-assisted extraction and thin-layer chromatography)、PA-m6 A-Seq(photo-crosslinking-assisted m6 A sequencing)、mi-CLIP-Seq(m6 A individual-nucleotide resolution crosslinking and immunoprecipitation sequencing)和m6 A-LAIC-Seq(m6 A level and isoform characterization sequencing)等方法来检测m6 A的分布[7,14-17]。如表1所示,本文对这些方法进行了简单描述,并分析了其存在的优缺点。通过试验的方法来鉴定m6 A位点既费时费力,又需要大量资金投入。因此,急需开发一些算法或程序来系统的分析鉴定m6 A位点。例如RFAthM6A、m6aViewer、TargetM6A、SRAMP、RNA-MethylPred、iRNA-Methyl和pRNAm-PC等算法或程序已被应用于动植物的m6 A位点预测[18-24]。这些算法或程序的使用将有助于更加准确、快速鉴定m6 A位点,以期更好的解释其生物学功能。
| 图表编号 | XD0044833000 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.01.01 |
| 作者 | 卢曾奎、张利平、李青、刘恩民、杜立新、储明星、魏彩虹 |
| 绘制单位 | 中国农业科学院北京畜牧兽医研究所、甘肃农业大学动物科学技术学院、甘肃农业大学动物科学技术学院、中国农业科学院北京畜牧兽医研究所、中国农业科学院北京畜牧兽医研究所、中国农业科学院北京畜牧兽医研究所、中国农业科学院北京畜牧兽医研究所、中国农业科学院北京畜牧兽医研究所 |
| 更多格式 | 高清、无水印(增值服务) |