《表2 根部真菌-植物互作网络结构特性指数》
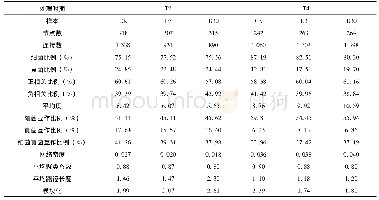
Patefield’s零模型(Patefild’s null model)算法随机分配真菌与植物的互作次数(Patefield,1981),同时确保每种真菌、植物的相互作用总次数与真实网络相同。数字加粗表示网络指数观察值显著高于/低于零模型预测值。
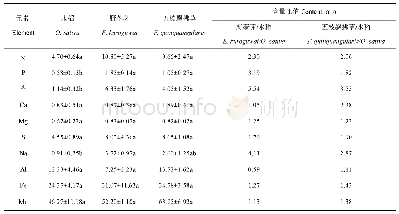
用于网络特征分析的3个互作矩阵为:根部真菌–植物(真菌420×植物8)、AM真菌–植物(真菌40×植物8)、ECM真菌–植物(真菌48×植物8)。网络的嵌套值在AM互作网络中最高(NODF=68.39),显著高于零模型预测值(NODFnull=60.70,P=0.025);在ECM互作网络中最低(NODF=26.06),显著低于零模型预测值(NODFnull=58.36,P=0.000);而根部真菌–植物互作网络的嵌套值介于二者之间(NODF=32.59),显著高于零模型预测值(NODFnull=9.40,P=0.000)(表2,图4) 。对于加权的嵌套性指数WNODF(weighted NODF),除了AM互作网络外,其他两类网络表现出与NODF相似的特征。
| 图表编号 | XD0041568500 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.03.01 |
| 作者 | 杨思琪、张琪、宋希强、王健、李意德、许涵、郭守玉、丁琼 |
| 绘制单位 | 海南大学热带农林学院环南海陆域生物多样性研究中心、海南大学热带农林学院环南海陆域生物多样性研究中心、海南大学热带农林学院环南海陆域生物多样性研究中心、海南大学热带农林学院环南海陆域生物多样性研究中心、中国林业科学研究院热带林业研究所、中国林业科学研究院热带林业研究所、中国科学院微生物研究所真菌学国家重点实验室、海南大学热带农林学院环南海陆域生物多样性研究中心 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式