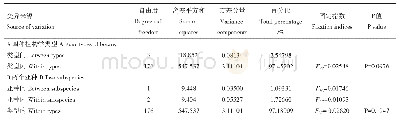
《表2 前鳞种群结构的分子方差分析及SAMOVA分析Tab.2 AMOVA and SAMOVA analysis of Liza affinis》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《基于线粒体COⅠ序列的中国前鳞(鱼夋)遗传多样性分析》
在AMOVA分析(表2)中,按照上文的群体划分进行分析:东兴、平潭、闸坡、广东、洞头等5个群体间的Fst=-0.0221~0.0141(P>0.05),组间的遗传变异比例达-0.26%。东海群体与南海群体间的Fst=0.0020(P>0.05),组间的遗传变异比例达0.46%。东兴、广东、东海3个群体间的Fst=-0.0159~0.0141(P>0.05),组间的遗传变异比例为-0.6%。上述各类划分中,群体间的Fs t值皆小于0.05,均表现出不显著水平(P>0.05),且组间的遗传变异比例均属于极低水平,表明中国前鳞群体在各个地区、海域、海峡间没有明显的遗传分化,遗传变异主要集中在群体中的个体间。而SAMOVA分析(表2)中,82条序列被软件自动划分为2组,即洞头群体独立归为1组,其余群体为1组,测得组间的变异比例只有2.87%,同样表明组间分化程度很低,与Fst及AMOVA分析的结果相吻合。
| 图表编号 | XD0034463300 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.02.01 |
| 作者 | 黄镇宇、章群、卢丽锋、周琪、唐楚林 |
| 绘制单位 | 暨南大学生态学系、暨南大学生态学系、暨南大学生态学系、暨南大学生态学系、暨南大学生态学系 |
| 更多格式 | 高清、无水印(增值服务) |