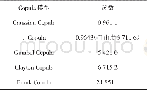
《表3 支位点模型下似然率值和参数估计Table 3 Likelihood values and parameter estimates under the branch-site models》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《花斑裸鲤Epo和Epor基因克隆及其低氧诱导的mRNA表达》
p0表示净化选择的比例,p1表示中性选择的比例,p2+p3表示正选择的比例;ω0、ω1和ω2分别为确定净化选择、中性选择和正向选择时的非同义替换与同义替换比值。p0 is the proportion of purifying selection;p1 is the proportion of neutral selection;p2+p3 is the proportion
系统发育分析结果显示,Epo和Epor基因进化树与已报道的物种进化树基本一致(Saitoh et al.2006),因此在选择压力分析中将Epo基因进化树作为输入树(input tree)。支-特异模型中自由比率分析结果表明,软刺裸鲤及其祖先支(支6,图3)Epo基因具有明显不同的进化速率(ω>1),而在花斑裸鲤支中并未检测到正向选择(ω=0.38)。同时,在所有硬骨鱼类的Epor基因中均未检测到进化速率较高的支系(图3)。为了进一步澄清特定支系Epo基因是否发生了特定位点的正向选择,本研究又进行了支-位点模型分析。结果表明,当花斑裸鲤作为前景支,而将其他物种作为背景支时,在花斑裸鲤Epo和Epor基因中均未检测到正向选择位点(ω2=1,ω2表示前景支相对于背景支的非同义替换与同义替换比值)(表3) 。然而将花斑裸鲤和软刺裸鲤的共同祖先支(支6,图3)作为前景支,而将其他物种作为背景支时,祖先支Epo进化速率较高于其他支(ω2=1.54,ω2花斑裸鲤和软刺裸鲤的共同祖先支相对于背景支的非同义替换与同义替换比值),但是似然比检验法显示差异不显著(2??=1.1,df=1,P=0.38);同样,花斑裸鲤和软刺裸鲤的共同祖先支Epor基因中也未检测到正向选择位点(ω2=1)(表3) 。
| 图表编号 | XD002342300 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2018.04.20 |
| 作者 | 赵永丽、吴蓉蓉、晁燕、陈祺昌、夏明哲、祁得林 |
| 绘制单位 | 青海大学省部共建三江源生态与高原农牧业国家重点实验室、青海大学生态环境工程学院、青海大学省部共建三江源生态与高原农牧业国家重点实验室、青海大学生态环境工程学院、青海大学农牧学院动物科学系、青海大学省部共建三江源生态与高原农牧业国家重点实验室、青海大学生态环境工程学院、青海大学省部共建三江源生态与高原农牧业国家重点实验室、青海大学省部共建三江源生态与高原农牧业国家重点实验室、青海大学农牧学院动物科学系 |
| 更多格式 | 高清、无水印(增值服务) |
查看“表3 支位点模型下似然率值和参数估计Table 3 Likelihood values and parameter estimates under the branch-site models”的人还看了
-

- Table 2–Estimated parameters, SSR and AIC values for the models, when membranes were loaded with 29 wt%Prg.
-
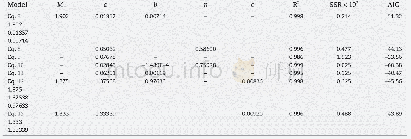
- Table 1–Estimated parameters, SSR and AIC values for the models, when membranes were loaded with 23 wt%Prg.
-
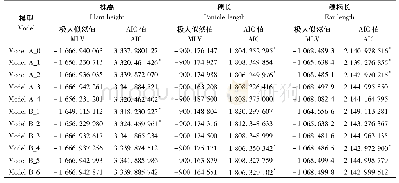
- 表3 高粱株高、穗长、穗柄长遗传模型的极大似然值和AIC值Tab.3 Maximum likelihood value and AIC values of genetic model of plant height, panicle len