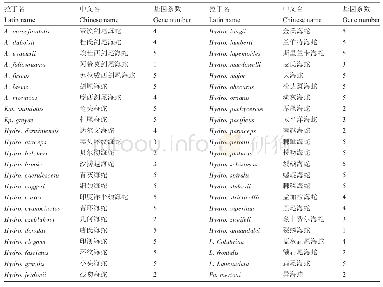
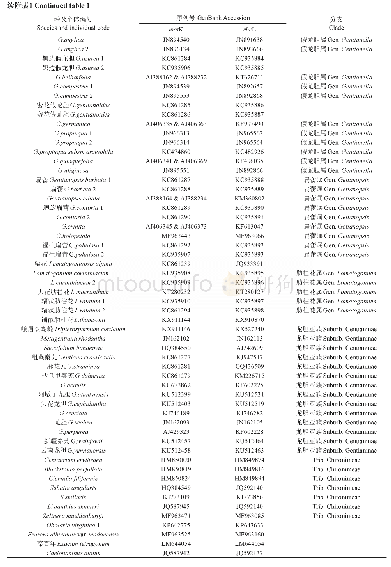
《表2 艾纳香属植物的ITS2序列(来源于Gen Bank)》
将测序峰图翻译成碱基序列,再利用Codon Code Aligner进行碱基Q值的质量评估,碱基序列两端的Q值较低,则删除,将反向序列翻译成正向序列,进行序列拼接。采用Keller (2009)基于隐马尔可夫模型的HMMer注释方法去除两端5.8S和28S区段即可获得ITS2间隔区序列。将拼接序列(表2)在Gen Bank数据库中的BLAST校对,确认该序列的种属是否正确。校对无误后,利用MEGA 7.0将其序列与所下载的近缘种和伪品的序列进行多重对比,分析变异位点,以Kimura 2-Parameter (K2P)模型计算种内、种间遗传距离,采用邻接法(neighbor-joining,NJ)构建系统聚类树。
| 图表编号 | XD00189146400 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.11.28 |
| 作者 | 魏妮娜、牛宪立、张青峰、邵敏 |
| 绘制单位 | 遵义医科大学珠海校区、遵义医科大学珠海校区、遵义医科大学珠海校区、遵义医科大学珠海校区 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式