《表4 蒙古扁桃17个居群的分子变异分析》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《基于叶绿体DNA非编码序列的蒙古扁桃谱系地理学研究》
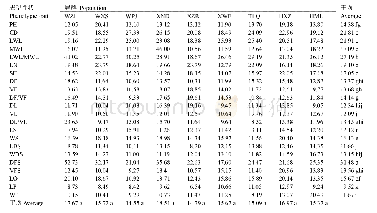
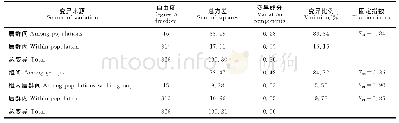
根据单倍型地理分布位置及Network单倍型网络关系(图2),把蒙古扁桃整个地理分布区分为东部居群组(P1~P9)与西部居群组(P10~P17)。利用ARLEQUIN软件包对蒙古扁桃居群进行分子变异分析(AMOVA),结果(表4)表明蒙古扁桃cpDNAtrnH-psbA序列的遗传变异居群间显著大于居群内,居群间的遗传变异为83.84%,居群内遗传变异为16.16%;对于东,西居群组而言,有84.72%的遗传变异来源于组间,5.55%的遗传变异来源于组内居群间,9.73%的遗传变异来源于居群内;总的居群间及东、西居群组间的Fst值为分别为0.84及0.90(P<0.001,1 000次重复的显著性检验)(表4) 。假设该序列变异处于漂变-迁移平衡(drift-migration equilibrium),则基于Fst值估算出物种水平上居群间的平均基因流值(Nm)为0.10。
| 图表编号 | XD0015676400 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2018.09.01 |
| 作者 | 段义忠、白春梅、段春燕、申烨华 |
| 绘制单位 | 榆林学院陕西省陕北矿区生态修复重点实验室、西北大学化学与材料学院、西北大学化学与材料学院、西北大学化学与材料学院、西北大学化学与材料学院 |
| 更多格式 | 高清、无水印(增值服务) |