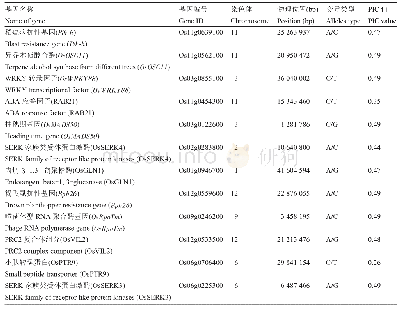
《表1 精简后可用于鉴定117份种质资源的一套12个核心SNP标记信息》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《基于全基因组SNP高效鉴定水稻种质资源并构建指纹图谱》
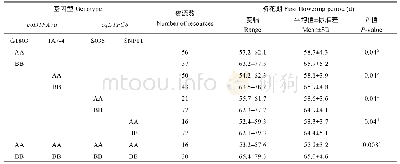
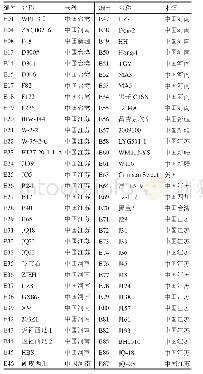
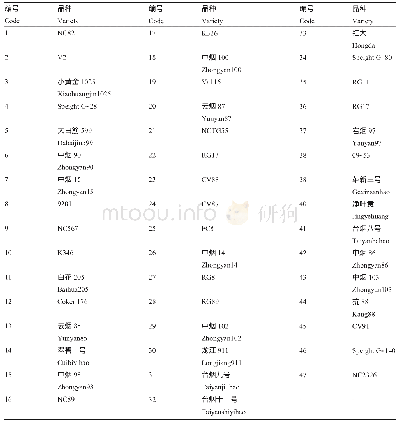
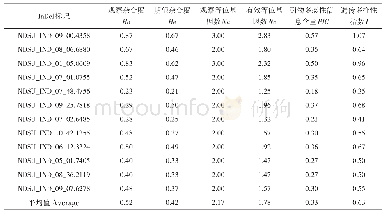
本研究通过新方法筛选出的12个核心SNP标记分别分布在稻瘟病抗性基因、异乔木萜醇合酶、WRKY转录因子等7条染色体上的12个基因上。对位于基因内的28 562个SNP的PIC值进行分布统计(图1),并计算的12个SNP标记的PIC值(表1)。结果表明基因内的28 562个SNP的多态性有高有低,但有大量SNP具有高多态性(35.1%的SNP标记PIC>0.4,23.2%的SNP标记PIC>0.45)。筛选出来的12个SNP标记83%具有高多态性(PIC>0.45),但也有2个SNP的多态性较低,分别为0.35和0.26。说明基因上有大量高多态性的SNP,筛选出来的12个SNP标记也有很大一部分是高多态性的,但是低多态性的SNP标记对于指纹图谱的构建也有一定价值的。
| 图表编号 | XD00146693300 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.09.28 |
| 作者 | 李梓榕、袁雄、陈叶、郑兴飞、胡中立、李兰芝 |
| 绘制单位 | 湖南农业大学农业大数据分析与决策工程技术研究中心、湖南农业大学农业大数据分析与决策工程技术研究中心、湖南农业大学农业大数据分析与决策工程技术研究中心、武汉大学杂交水稻国家重点实验室、武汉大学杂交水稻国家重点实验室、湖南农业大学农业大数据分析与决策工程技术研究中心、湖南杂交水稻研究中心杂交水稻国家重点实验室 |
| 更多格式 | 高清、无水印(增值服务) |