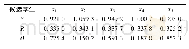《表2基于No r m f\n\t\t\t\t\ti n d\n\t\t\t\t\te r软件分析候选m i RA N内参基因的稳定性Ta b l e2Th e s t a b
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《核桃(Juglans regia L.)MicroRNA实时荧光定量RT-PCR内参基因的筛选》
RT-qPCR因其快速、灵敏、特异等优势已被广泛用于基因的表达分析。选择合适的内参基因对数据进行标准化是得到正确结果的关键(尹冬梅等,2017)。理想内参基因在不同基因型、不同组织和不同实验条件下均能稳定表达。然而,大量研究表明并没有绝对稳定的基因(Kulcheski et al.,2010)。目前,关于miRNA定量分析的内参筛选多集中于医学及动物研究领域。在植物miRNA的定量分析中,5S rRNA(Qu et al.,2016)、5.8S rRNA(Sun et al.,2017)、18S r-RNA(Li et al.,2016)、U6 snRNA(Bai et al.,2016)等常用作内参基因。本研究中,U6 sn RNA的扩增效率E为211.1%,相关系数R2为0.600,不适合作为内参基因。这与小麦中U6 snRNA(E:458.6%;R2:0.648)不适合作为内参的结果一致(Feng et al.,2012)。在龙眼中,U6 snRNA稳定性最差(Lin and Lai,2013),而在柑橘中,U6 snRNA稳定性排名第二(Kou et al.,2012)。由此可见,U6 sn RNA在不同物种中表达量是不稳定的。采用RT-qPCR分析了特异性较强的8个候选内参基因在不同分化期花芽与叶芽、不同组织及品种中的表达量,并利用geNorm、Normfinder和RefFinder软件对其稳定性进行了评价。结果表明,没有一个候选内参基因在各实验中均稳定表达。在不同分化期花芽中,geNorm显示jre-miR159c为最稳定的单基因,jre-miR394a和jre-miR159a为最稳定的组合。V3/4小于0.15(图4E),需要三个内参基因,即jre-miR394a、jre-miR159a和jre-miR159c。Normfinder显示稳定性排名前三分别是jre-miR394a、jre-miR159a和jre-miR159c。RefFinder结果与Normfinder完全一致。在不同分化期叶芽中,geNorm显示最稳定的单基因为jre-miRn7,最稳定的组合为5.8S rRNA和jre-miRn3。V2/3小于0.15(图4E),最适的内参数目为2个,即5.8S r RNA和jre-miRn3。Normfinder则显示jre-miR167d最稳定,jre-miR159c、jre-miRn7和jre-miR394a次之。RefFinder显示5.8S rRNA最稳定,jre-miRn3、jre-miR394a和jre-miRn7次之。在生物胁迫和非生物胁迫下的小麦中,最适的内参基因为miR167和miR159(Feng et al.,2012)。在干旱胁迫下的大豆叶中,最适的单个内参基因为miR167a,最适的内参组合为miR167a与miR1520d(刘伟灿等,2016)。在梨中,miR167d稳定性一般,不同软件分析的结果也有差异(马鑫瑞等,2018)。表明miR167在不同物种中稳定性并非稳定不变。在进行miRNA定量分析时,应根据不同的物种或同一物种的不同组织筛选最适的内参基因。在不同组织和不同品种中,三种软件分析的最稳定的基因基本一致,只是排序有所差异。这种差异可能是由不同软件的统计算法导致的。此外,所有的V值均大于0.15,且随着内参数目n的增加,V值并没有太大变化(图4E)。在小麦不同组织和基因型(Feng et al.,2012)、石蒜属植物不同花期和不同组织中(蒋婷婷等,2015),所有V值也大于0.15。其原因可能是不同组织、不同品种和基因型跨度较大,样本数目不够或检测的内参基因数目有限。geNorm操作说明也提到Vn/n+1并非一定要设置为0.15(Vandesompele et al.,2002)。因此,在不同实验条件下,应结合不同软件筛选出最适的内参基因。
| 图表编号 | XD0031234400 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.04.14 |
| 作者 | 周丽、全绍文、马丽、徐航、牛建新 |
| 绘制单位 | 石河子大学农学院园艺系特色果蔬栽培生理与种质资源利用兵团重点实验室、石河子大学农学院园艺系特色果蔬栽培生理与种质资源利用兵团重点实验室、石河子大学农学院园艺系特色果蔬栽培生理与种质资源利用兵团重点实验室、石河子大学农学院园艺系特色果蔬栽培生理与种质资源利用兵团重点实验室、石河子大学农学院园艺系特色果蔬栽培生理与种质资源利用兵团重点实验室 |
| 更多格式 | 高清、无水印(增值服务) |