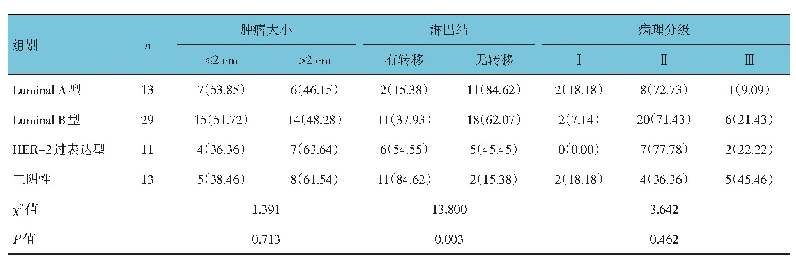
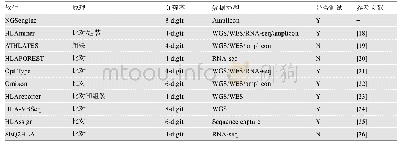
《表1 7 种HLA分型软件比较》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《三代测序与靶向捕获技术联用进行高分辨HLA基因分型及MHC区域单倍体型精细鉴定》
WGS:全基因组数据;WES:全外显子组数据;RNA-seq:转录组数据;Amplicon:基因扩增子数据;Y:对应软件已测试;N:对应软件未测试。
利用包括全基因组、全外显子组、转录组及HLA基因扩增子数据在内的多种数据类型进行HLA分型的各种学术和商业化软件已被广泛使用(表1)。为评估分型软件的准确性以便从中选择最佳的分型软件用于与后续三代数据分型结果的比较,比较了7种不同的HLA分型软件对基于全长扩增子二代测序数据的分析性能。为了确保分型结果的可靠性,不论是Illumina Miseq还是PacBio RSII均保证了足够的数据量。由于二代数据存在一定偏好,因此其覆盖度会存在不均一的情况,不同基因的覆盖度有一定的差异(图1),但是总体而言,其95%以上的区域覆盖度会大于200×。而三代数据的覆盖度比二代数据更高(图1)。通过比较,7种软件对二代数据的分型结果展现出较大差异。从总体上来看,NGSengine对classⅠ和classⅡ基因的分型结果敏感度都较高,结果与血清学鉴定结果吻合。而其他6种分型软件仅对classⅠ类基因的分型均较为准确,但对classⅡ类基因则不敏感,部分软件甚至不能给出分型结果。例如,HLAssgin和HLAreporter只有2个(2/12)和0个样本(0/12)预测到了DPA1基因分型。而HLAminer和Omixon对classⅠ基因的分型结果的判定与NGSengine/血清学结果相比有很大的差异。由于HCC27样本的HLA基因分型结果经过了血清学结果及MHC区域捕获测序结果的双重验证,故以该样本作为示例(表2),NGSengine对classⅠ和classⅡ基因的分型结果与血清学鉴定结果以及MHC区域捕获测序结果高度吻合。但是HLAssgin和HLAreporter无法预测到DPA1的基因分型,而HLAminer和Omixon对DPB1、DQB1、DRB1等classⅡ基因的分型结果判定错误率较高(表2)。
| 图表编号 | XD0045287100 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.04.01 |
| 作者 | 陈佳、舒明月、里进、付爱思、杨帆、王邹、李一荣、邓子新、刘天罡 |
| 绘制单位 | 武汉大学药学院组合生物合成与新药发现教育部重点实验室、武汉大学药学院组合生物合成与新药发现教育部重点实验室、武汉大学中南医院检验医学中心、武汉大学药学院组合生物合成与新药发现教育部重点实验室、武汉生物技术研究院公共技术服务平台、武汉生物技术研究院公共技术服务平台、武汉大学中南医院检验医学中心、武汉大学药学院组合生物合成与新药发现教育部重点实验室、武汉大学药学院组合生物合成与新药发现教育部重点实验室 |
| 更多格式 | 高清、无水印(增值服务) |