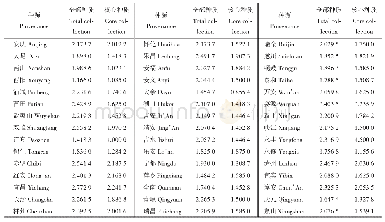
《表3 矮拟帽贝的遗传多样性 (标准差) Tab.3 Genetic diversity of P.pygmaea (standard deviation)》
注:JZ:锦州;ZH:庄河;DL:大连;YT:烟台;WH:威海;QD:青岛;LYG:连云港;ZS:舟山;XM:厦门;ST:汕头;MM:茂名;ZJ:湛江;BH:北海Note:JZ:Jinzhou;ZH:Zhuanghe;DL:Dalian;YT:Yantai;WH:Weihai;QD:Qingdao;LYG:Lianyungang;ZS:Zhoushan;XM:Xiamen;ST:Shantou;MM:Maoming;ZJ:Zhanjiang;BH:Be
137个矮拟帽贝个体线粒体DNA COI部分序列(601bp)碱基含量:A:24.10%、T:37.27%、C:16.53%、G:22.10%,共发现217个多态位点,转换颠换比为1.25,无插入/缺失,界定30个单倍型,占样本总数的21.89%。AMOVA结果显示,矮拟帽贝遗传变异主要来源于种群间(89.57%,P<0.001)。各种群遗传多样性比较显示,汕头(ST)种群呈现较高的遗传多样性,威海(WH)种群呈现较低的遗传多样性(表3)。对COI分子标记的遗传多样性指数进行图示法和正态分布检验,所有遗传多样性指数都服从正态分布。与样本量之间的相关性分析结果显示,各多样性指数均不受个体数量的影响(P>0.05),正态分布检验和相关性分析的结果说明此数据可以用于表示各地理种群的遗传多样性高低。
| 图表编号 | XD0034277900 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.01.01 |
| 作者 | 李媛媛、王珊珊、郭晓雨、崔宇通、王云慧、王儒晓、李钊霞、蔡建平、张运发、曲江勇 |
| 绘制单位 | 烟台大学生命科学学院、烟台大学生命科学学院、烟台大学生命科学学院、烟台大学生命科学学院、烟台大学数学与信息科学学院、烟台大学生命科学学院、烟台大学生命科学学院、烟台大学生命科学学院、烟台大学生命科学学院、烟台大学生命科学学院 |
| 更多格式 | 高清、无水印(增值服务) |
查看“表3 矮拟帽贝的遗传多样性 (标准差) Tab.3 Genetic diversity of P.pygmaea (standard deviation)”的人还看了
-

- 表3 角倍蚜干雌和越冬若蚜的遗传多样性分析Tab.3 Genetic diversity of the fundatrigeniae and overwintering larvae of S.chinensis
-
- 表3 全部种质与核心种质各种源遗传多样性指数Tab.3 Index of genetic diversity in entire germplasm and core collection of different provenances
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式





