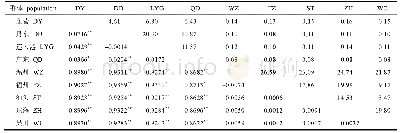
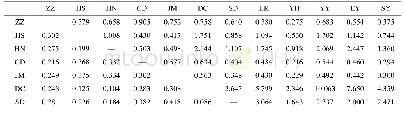
《表4 5个野生榧树居群成对遗传分化系数Fst(上三角)与基因流Nm(下三角)》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《基于EST-SSR标记的野生榧树居群遗传多样性分析》
注:“*”表示Popgene计算的P值
依据36对引物在5个野生榧树居群的142个单株中的扩增结果,统计后利用Popgene软件计算得到居群遗传分化指标。36个SSR位点的F统计与基因流分析结果(表3)显示,居群近交系数(Fis)的变化范围从–0.210 1(ZAFU-19)至0.980 7(ZAFU-30),平均近交系数为0.398 1。遗传分化系数(Fst)的变化范围从0.085 6(ZAFU-13)至0.660 4(ZAFU-24),平均遗传分化系数为0.261 9。居群基因流(Nm)的变化范围从0.213 1(ZAFU-35)至3.395 7(ZAFU-31),所有位点平均基因流为0.704 7,其中,ZAFU-13,ZAFU-22和ZAFU-313个位点Nm大于2。5个野生居群成对分析结果(表4)显示,居群两两之间的Fst均大于0.15,居群间遗传分化明显,除HS-SZ居群和SZ-SY居群,居群间的Nm大于1。利用GenALEx软件对居群进行分子方差分析(AMOVA)的结果(表5)显示,5个榧树居群的遗传变异主要来于居群内(72%)。
| 图表编号 | XD00144832400 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.03.30 |
| 作者 | 陈焘、李杰峰、张迟、高樟贵、金航标、戴文圣、唐海英、张敏 |
| 绘制单位 | 浙江农林大学亚热带森林培育国家重点实验室、松阳县自然资源和规划局、浙江农林大学亚热带森林培育国家重点实验室、景宁嘉树农业科技开发有限公司、松阳县自然资源和规划局、诸暨市自然资源和规划局、浙江农林大学亚热带森林培育国家重点实验室、磐安县自然资源和规划局、浙江农林大学亚热带森林培育国家重点实验室、景宁嘉树农业科技开发有限公司 |
| 更多格式 | 高清、无水印(增值服务) |