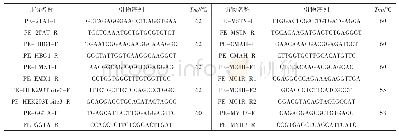
《表1 引物序列信息:利用CRISPR/Cas9和piggyBac实现果蝇基因组无缝编辑》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《利用CRISPR/Cas9和piggyBac实现果蝇基因组无缝编辑》
斜体下划线分别为酶切位点Eco RⅠ和Acc65Ⅰ序列,仅下划线为同义突变碱基,粗体下划线为点突变碱基,粗体为TTAA序列。
将pGX-attP载体(载体详情见参考文献[19])上两个多克隆位点之间的attP-hsp70-white(从Acc65Ⅰ位点至SpeⅠ位点)替换为两端带有piggyBac转座酶识别位点ITRs的Ds Red标记基因,获得载体pGX-DsRed。在目标突变位点附近寻找TTAA位点作为标记基因插入位置,在此序列前后各约1.2 kb和1.1 kb分别设计5′同源臂和3′同源臂,同源臂扩增使用的PCR酶为Phanta高保真酶(南京诺唯赞公司)。扩增5′同源臂的引物为FW3、RV3、FW4和RV4(序列见表1),为了引入突变碱基,本研究将5′同源臂分成2部分进行扩增。由于供体DNA质粒中包含sgRNA序列,为防止供体质粒注入果蝇胚胎后被Cas9切割,本研究对这部分序列中的3个氨基酸进行了同义突变(GGT→GGA,AAT→AAC,ATC→ATT),同时扩增过程中还在FW3引物中引入了点突变(TAT→TGT),编码氨基酸由酪氨酸变成半胱氨酸。3′同源臂扩增引物为FW4和RV4(序列见表1)。为保证DsRed标记在后续实验过程中顺利移除,5′同源臂的3′端和3′同源臂的5′端均带有TTAA序列,这样经piggyBac转座酶处理后才能得到无缝效果。扩增得到的片段使用多片段同源重组酶(南京诺唯赞公司)进行连接。
| 图表编号 | XD0060412500 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.05.01 |
| 作者 | 王珏、黄娟、许蕊 |
| 绘制单位 | 南京医科大学基础医学院医学遗传学系、南京医科大学基础医学院医学遗传学系、南京医科大学基础医学院医学遗传学系 |
| 更多格式 | 高清、无水印(增值服务) |