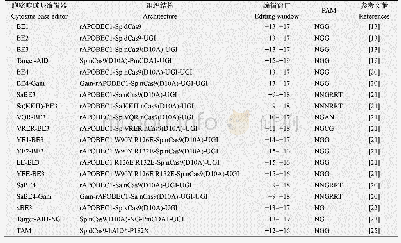
《表2 胞嘧啶碱基编辑器的结构和编辑窗口》
当编辑窗口存在多个胞嘧啶时,胞嘧啶核苷脱氨基酶会使其它非目标胞嘧啶也发生突变。为了减少这种情况的出现,Kim等又在BE3的基础上开发了编辑窗口更小的碱基编辑器,他们将BE3中的APOBEC1进行点突变,筛选到的突变体使编辑窗口减小到1-2 bp,从而显著增加了碱基编辑的精确性[21]。此外,现有碱基编辑器在识别编辑单链DNA上的胞嘧啶时需依赖PAM序列的存在,但往往很多编辑位点的附近没有合适的PAM序列。为了解决这一问题,Kim等将BE3中的nCas9(D10A)引入各种点突变,或替换为来源于金黄色葡萄球菌的Sa nCas9(D10A),改良版BE3识别的PAM序列更加多样,进而增加了DNA上的可编辑位点(约2.5倍)[21]。Hu等采用噬菌体辅助的连续进化(PACE)技术,筛选到可以识别更多PAM序列的Cas9,命名为x Cas9[22]。将BE3中的n Cas9(D10A)替换为xCas9(D10A)构建的xBE3,可以有效识别并编辑附近存在PAM序列为NGN、GAA、GAT的胞嘧啶碱基。x Cas9不仅可以识别更多的PAM,而且它的使用还进一步降低了编辑的脱靶率[22]。Nishimasu等进行了类似的研究工作,他们采用理性设计的方法获得了一种Cas9突变体,命名为NG-Cas9[23]。基于NG-d Cas9的编辑器可以有效编辑某些PAM序列为NG附近的胞嘧啶位点[23]。最近,研究发现DNA的甲基化修饰会降低BE3的编辑效率,Wang等将鼠源的APOBEC1替换为来自人类的APOBEC3A(hA3A),构建的hA3A-BE3可以明显减少DNA甲基化对编辑效率的影响,实现对甲基化胞嘧啶的有效编辑[24]。Ma等将Human activation-induced deaminase(hAID)的C端去除得到AIDx,将d Cas9与AIDx进行融合开发了一种Targeted AID-mediated mutagenesis(TAM),这种方法可以将胞嘧啶转化为其它3种碱基[25]。此外,Gehrke等开发的eA3A-BE3可以在不改变编辑窗口的情况下,优先编辑“TC”位点中的胞嘧啶,这种方法使碱基编辑更加精准[26](表2)。以上研究工作,尤其是哈佛大学David R.Liu实验室的工作[13,20-22],一方面显著提升了CBE的效率以及精确性,另一方面也大幅度增加了基因组上的可编辑位点,这些努力为其应用于哺乳动物、植物细胞基因组的精准编辑打下了良好的基础。
| 图表编号 | XD0043794500 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.02.20 |
| 作者 | 赵亚伟、姜卫红、邓子新、汪志军、芦银华 |
| 绘制单位 | 上海师范大学生命科学学院、上海交通大学生命科学技术学院微生物代谢国家重点实验室、中国科学院分子植物科学卓越创新中心、植物生理生态研究所合成生物学重点实验室、上海交通大学生命科学技术学院微生物代谢国家重点实验室、上海交通大学生命科学技术学院微生物代谢国家重点实验室、上海师范大学生命科学学院 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式