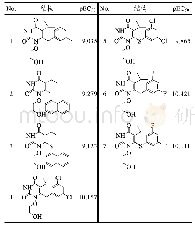
《表7 基于QSAR模型设计的分子结构和预测值》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《HEPT类HIV-1逆转录酶衍生物的CoMFA、CoMSIA及HQSAR分析》
分子场的三维等势图由于覆盖的区域过大,不能准确定位需要修改基团的位置;HQSAR模型的色码贡献图能够准确定位分子场的取代活性位点,但不能判断取代基团的性质,因此,CoMFA、CoMSIA模型和HQSAR模型的结合可以准确确定目标分子的活性取代位点。综合分析可知,立体场和疏水场是影响分子活性的主要因素,因此从立体场和疏水场出发,对分子进行修饰。HQSAR模型显示,R2取代基为3,5-二甲基苯硫,色码图显示为橙红色,对分子活性不利,其余取代基处色码图为白色,对分子活性无特别贡献;分子场三维等势图显示,在R2取代基位置处改造空间较大,所以应在R2位置处引入大基团的亲水性取代基。表7是根据QSAR模型设计的理论上具有高活性的7个分子。
| 图表编号 | XD0035913300 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.01.15 |
| 作者 | 秦尚尚 |
| 绘制单位 | 陕西科技大学化学与化工学院、教育部轻化工助剂化学与技术重点实验室 |
| 更多格式 | 高清、无水印(增值服务) |