《表1 高通量测序数据质量控制统计》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《基因组测序技术解析耐除草剂转基因水稻G2-7的分子特征》
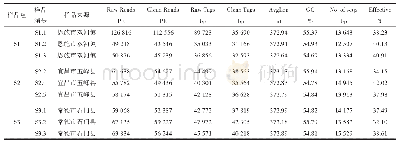
Q20、Q30分别代表Phred数值大于20、30的碱基占总体碱基的百分比,表示的碱基正确识别率为99.0%和99.9%。
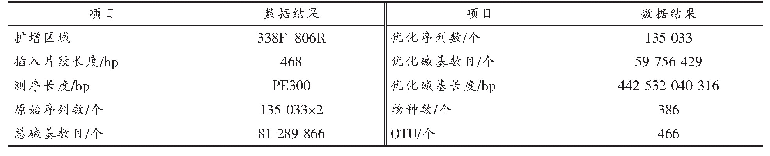
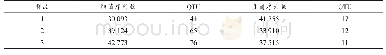
Illumina Nova Seq 6000高通量测序得到的原始图像数据文件经CASAVA碱基识别(base calling)分析转化为原始测序序列(raw bases)。对获得的原始测序数据进行质量控制,过滤掉带接头(adapter)的读序、单端测序读序中N数量超过此读序长度比例10%的读序、及单端测序读序中含有的低质量(Q≤5)碱基数超过该条读序长度比例50%的等低质量读序,获得Clean Bases和Clean Reads。本研究获得样品的原始测序量为41.16~47.13 G,有效读序为274,371,776~314,171,200,Q30大于90%,说明获得的测序数据丰富可靠(表1)。
| 图表编号 | XD00226868300 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.11.12 |
| 作者 | 马硕、焦悦、杨江涛、王旭静、王志兴 |
| 绘制单位 | 中国农业科学院生物技术研究所、农业农村部农业转基因生物安全评价(分子)重点实验室、农业农村部科技发展中心、中国农业科学院生物技术研究所、农业农村部农业转基因生物安全评价(分子)重点实验室、中国农业科学院生物技术研究所、农业农村部农业转基因生物安全评价(分子)重点实验室、中国农业科学院生物技术研究所、农业农村部农业转基因生物安全评价(分子)重点实验室 |
| 更多格式 | 高清、无水印(增值服务) |