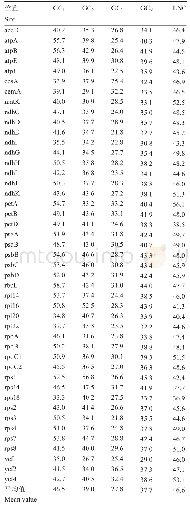
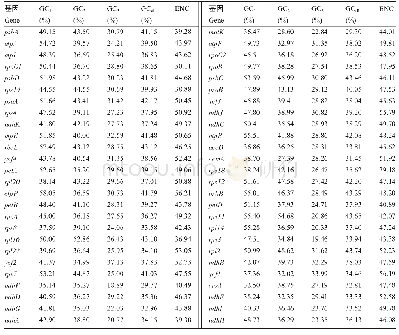
《表1 5个非洲菊CAD基因位于编码区不同位置的密码子GC含量及ENC和CAI值》
从非洲菊转录组数据中筛选获得5条具有完整CDS的CAD基因,分别命名为CAD1、CAD2、CAD3、CAD4、CAD5。通过CodonW软件计算它们的ENC和CAI值,利用EMBOSS在线分析得它们的总GC含量以及位于密码子不同位置的GC含量,整理得表1。由表1可以看出,5个非洲菊CAD基因的ENC值相差不大,介于53.37~58.07,平均值为55.77,说明非洲菊CAD基因密码子的使用偏好性较弱。从CAI指数看,非洲菊CAD基因CAI值介于0.191~0.225,平均值为0.210,远小于1,进一步说明了非洲菊CAD基因的密码子偏好性较弱。总GC含量为44.16%~50.23%,平均值为46.46%;GC1s为48.46%~55.71%,平均值为52.56%;GC2s为37.05%~40.67%,平均值为39.16%;GC3s为44.08%~57.94%,平均值为47.67%,由此推测非洲菊CAD基因家族编码氨基酸时使用较多的A或T,G或者C的偏好性较弱,且偏向于以A或T结尾。
| 图表编号 | XD0060993500 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.04.15 |
| 作者 | 王星淇、石小保、林芳柯、孙雪丽、刘范、田娜、郝向阳、程春振 |
| 绘制单位 | 福建农林大学园艺学院、福建农林大学园艺学院、福建农林大学园艺学院、福建农林大学园艺学院、福建农林大学园艺学院、福建农林大学园艺学院、福建农林大学园艺学院、福建农林大学园艺学院 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式