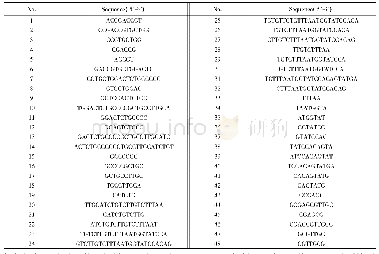
《Table 1Sequences for DNA aptamers》
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式
本系列图表出处文件名:随高清版一同展现
《Cε3-Cε4蛋白寡核苷酸适配子结合位点的预测与筛选》
使用MMB程序,设置碱基对序列和温度等参数,构建了46个PDB格式的DNA适配子发夹结构,其中序列2和序列24的发夹结构构建失败.这46个适配子发夹结构长度各不相等,短至5个碱基(序列5),长则可达26个碱基(序列14),但大部分均以短序列存在,以获取与蛋白作用的最短核心部位.利用PyMol软件将短发夹适配体的结构显示成Cartoon,图2显示了长度各不相同的短发夹适配体结构,序列4,18和21[图2(A),(C) 和(D)]为分别由6,9和11个碱基形成的较短发夹结构;序列10,22和29[图2(B),(E) 和(F)]为分别由24,18和18个碱基,通过2个碱基互补配对形成的1个大环状发夹结构.构建适配子3D结构是蛋白与核酸对接的关键[14],利用RNA composer程序[15]对适配子A1的发卡结构进行了预测[图2(G)~(L)],通过对比发现2种方法具有一定的相似性,MMB比RNA Composer更具灵活性,可以指定碱基配对等参数,允许分子的不同区域以不同的灵活性进行处理,能够人为地控制核酸的空间结构[16,17].
| 图表编号 | XD0031434500 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2019.01.10 |
| 作者 | 刘中成、刘世芳、张苏、杨艳蕾、李飞、张楠、袁欣、张艳芬 |
| 绘制单位 | 河北大学药学院、河北省药物质量分析控制实验室、河北大学药学院、河北省药物质量分析控制实验室、河北大学药学院、河北省药物质量分析控制实验室、河北大学药学院、河北省药物质量分析控制实验室、河北大学药学院、河北省药物质量分析控制实验室、河北大学药学院、河北省药物质量分析控制实验室、河北大学药学院、河北省药物质量分析控制实验室、河北省药物质量分析控制实验室、河北大学科学技术处 |
| 更多格式 | 高清、无水印(增值服务) |





