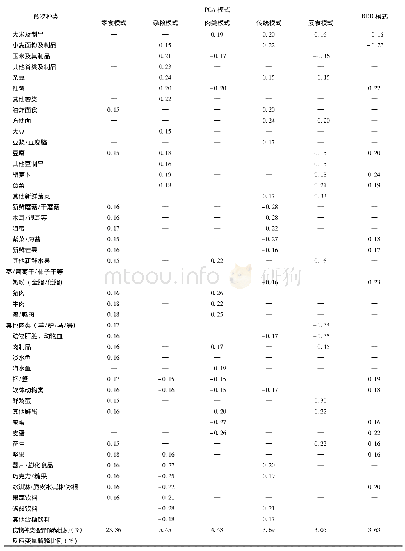
《表1 SFS-RF、LASSO和PCA方法的比较》
肿瘤基因表达具有多变量、高维度和高冗余的特点,为了获得有效且可靠的信息,LASSO回归和PCA已广泛运用于生物标志物的筛选[29~30]。根据TCGA数据库中下载的乳腺癌RNA数据,利用SFS-RF、LASSO和PCA方法筛选生物标志物,用ACC、SN、SP、MCC和ROC曲线衡量它们筛选生物标志物的分类精度。3种方法筛选出的生物标志物数量及分类精度见表1。表1直观地显示:在分类精度相近的情况下,SFS-RF、LASSO和PCA方法筛选出来的生物标志物数量依次为3、19和9,显然SFS-RF方法筛选出的生物标志物最少。在乳腺癌的研究中,通过本文提出的方法,只需检测3个基因的转录水平就能达到与其他方法(如LASSO回归和PCA)相近的识别精度。因此,SFS-RF方法在筛选生物标志物时具有更高的效率。
| 图表编号 | XD00138358500 严禁用于非法目的 |
|---|---|
| 绘制时间 | 2020.02.29 |
| 作者 | 朱东月、朱平 |
| 绘制单位 | 江南大学理学学院、江南大学理学学院 |
| 更多格式 | 高清、无水印(增值服务) |
 提示:宽带有限、当前游客访问压缩模式
提示:宽带有限、当前游客访问压缩模式